Introduction
La variabilité génétique1 du virus SARS-CoV-22 depuis le début de la pandémie a modifié plusieurs aspects épidémiologiques de sa circulation dans le monde. Dès le début de l’automne 2020, les virologues ont observé l’émergence de plusieurs « variants » issus de différents lignages du SARS-CoV-2 dans des zones géographiques distinctes. Ces variants étaient caractérisés par l’acquisition d’une nouvelle mutation ou, le plus souvent, d’une combinaison de mutations (substitutions amino-acidiques, délétions3 ou insertions) conférant un phénotype adaptatif aux variants correspondants (meilleure infectivité et/ou sensibilité diminuée aux anticorps neutralisants). Ces propriétés ont permis aux variants émergents (en particulier les variants α, β et γ) de se propager rapidement à de nombreuses autres régions du monde dès la fin de 2020 (Figure 1), quelques mois plus tard pour le variant δ et depuis novembre 2021 pour le variant Omicron. Les institutions de santé publique nationales et internationales (par exemple l’Organisation mondiale de la santé) se sont mobilisées pour mettre en place une stratégie d’analyse en temps réel de l’évolution de la circulation des variants et les classer en fonction de leur impact sur la santé publique. Ainsi, les capacités de surveillance génomique par séquençage du génome entier se sont renforcées : en France, le projet Emergen (Consortium pour la surveillance et la recherche sur les infections à pathogènes émergents via la génomique microbienne), coordonné par Santé publique France et l’ANRS Maladies infectieuses émergentes4, s’est organisé à partir du début de 2021 pour déployer un système de surveillance génomique des infections à SARS-CoV-2 couvrant l’ensemble du territoire national. La surveillance génomique française repose sur des enquêtes d’abord hebdomadaires puis bi-hebdomadaires fondées sur un séquençage aléatoire de SARS-CoV-2 isolés à partir d’échantillons d’origine respiratoire, avec pour objectif de définir la répartition des variants circulants et leur dynamique au cours du temps. En parallèle, des enquêtes ciblées (analyses de clusters, des réinfections, des cas graves) sont menées en coordination avec les agences régionales de santé et Santé publique France. L’ensemble des données des séquences est déposé dans la base de données virologique internationale GISAID (Global initiative on sharing avian influenza data5).
Les coronavirus, famille à laquelle appartient le SARS-CoV-2, sont des virus enveloppés dont le génome est une molécule d’ARN6 monocaténaire (ARN simple brin) d’une taille d’environ 30 kb, ce qui en fait le plus grand des génomes ARN de virus infectant l’homme. L’ARN génomique est protégé par un nucléocapside de structure tubulaire à symétrie hélicoïdale (formée par la protéine N) et par une protéine membranaire ou de matrice (protéine M). L’enveloppe virale porte à sa surface plusieurs protéines, dont une protéine de fusion de type I, la protéine « Spike » (protéine S), organisée en trimères et donnant un aspect en « spicules ». La partie externe de Spike (S1) porte le domaine de liaison au récepteur (RBD, pour receptor binding domain), qui permet la liaison du virus à son récepteur cellulaire, l’enzyme de conversion de l’angiotensine 2 (ACE27). Ce domaine porte la majorité des épitopes neutralisants. La partie C-terminale de la protéine Spike ou S2 assure la fusion de l’enveloppe virale et de la membrane cellulaire lors de l’entrée du virus dans la cellule cible. Cette fusion est amorcée par le clivage de la protéine Spike en ses deux sous-unités S1 et S2 par la protéase transmembranaire à sérine 2 (TMPRSS28).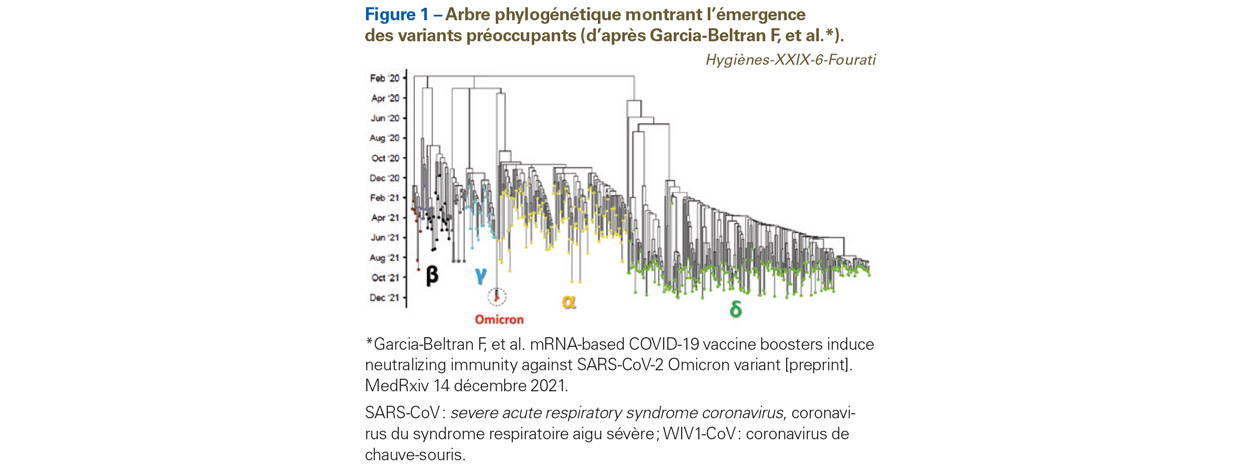
Mécanismes de la variabilité génétique du SARS-CoV-2
La diversité génétique du SARS-CoV-2 est la conséquence de plusieurs mécanismes : recombinaison entre différents coronavirus pouvant faire émerger des virus « mosaïques9 » (par exemple recombinant entre variants B.1.177 et α ou entre variants α et δ) ; mutations ponctuelles (substitutions, délétions ou insertions) dues aux erreurs de la polymérase virale –le taux de mutation est néanmoins inférieur à celui observé pour d’autres virus à ARN (tels que le virus influenza ou le virus de l’hépatite C), en lien avec l’existence d’une exoribonucléase virale (NSP1410) qui permet de corriger partiellement les erreurs introduites par la polymérase virale– ; ou édition génomique par des adénosines-désaminases cellulaires (Adar11), qui pourrait également contribuer à la variabilité génétique virale.
Évolution adaptative
Comme pour les autres virus à ARN, l’évolution adaptative du SARS-CoV-2 est avant tout la conséquence des erreurs générées par la polymérase virale au cours de la réplication des génomes à l’origine de l’apparition des substitutions, délétions ou insertions d’acides aminés au sein des protéines virales, qui sont ensuite sélectionnées sur la base des avantages sélectifs conférés au virus. Les variants les mieux adaptés à l’environnement réplicatif, à la fois chez le sujet infecté et au sein des populations dans lesquelles le virus se propage (transmissibilité, efficacité de la réplication virale, évasion immune, etc.), deviennent majoritaires au cours du temps. Ainsi, au début de la pandémie, alors que la plupart des populations humaines n’avaient pas été exposées au SARS-CoV-2, l’évolution adaptative a rapidement favorisé un variant porteur de la mutation D614G12 dans la protéine Spike ayant une transmissibilité accrue par rapport au variant « Wuhan » d’origine. Au fur et à mesure que la pandémie a progressé, plusieurs lignages distincts ont émergé puis diffusé dans le monde, de sorte que de nombreux variants circulent actuellement, ceux qui présentent un avantage de transmissibilité à un instant donné dans une population donnée devenant majoritaires. Par ailleurs, la population humaine acquiert au cours du temps une immunité vis-à-vis du virus, soit à la suite d’une infection (immunité naturelle), soit après une vaccination (immunité post-vaccinale). Cet environnement de plus en plus contraint favorise la sélection de mutations d’échappement (le plus souvent partiel) à la réponse immunitaire humorale. Sur le plan virologique, les mutations adaptatives concernent principalement (mais pas seulement) la protéine Spike, plus particulièrement les domaines de liaison au récepteur (RBD) et la région N-terminale (NTD13), les deux sites principaux ciblés par la grande majorité des anticorps neutralisants (Figure 2). Certaines mutations dans le RBD confèrent un avantage sélectif au virus en permettant d’améliorer l’interaction de Spike avec le récepteur cellulaire (N501Y, L452R), tandis que d’autres mutations en amont du site de clivage de Spike (P681H/R) permettraient d’améliorer le potentiel fusogène de la protéine Spike, facilitant la diffusion tissulaire. Plusieurs des mutations sélectionnées citées ci-dessus confèrent un avantage majeur au virus et ont ainsi été sélectionnées dans des lignages viraux indépendants (non monophylétiques) ayant émergé dans différentes régions du globe. Ce phénomène est appelé convergence évolutive. Ainsi, la mutation « prototype » E484K dans la protéine Spike est trouvée à la fois dans la séquence des variants préoccupants β (lignage B.1.351), dit variant « sud-africain », et γ (lignage P1), dit variant « brésilien ». Elle a également été sélectionnée au cours du temps par d’autres variants, y compris le variant α (lignage B.1.1.7), dit variant « britannique ». Cette mutation a également été décrite comme étant apparue au cours du temps lors d’infections persistantes chez des sujets immunodéprimés ou chez des sujets recevant une thérapie par anticorps monoclonaux. Au fur et à mesure, la forte circulation de certains variants dans plusieurs régions du monde favorise l’émergence de nouveaux sous-lignages porteurs de nouvelles substitutions ou délétions dans Spike permettant une meilleure adaptabilité virale. Ainsi, par exemple, de nouveaux sous-lignages du variant δ et γ (qui a longtemps circulé en Amérique du Sud) ont récemment émergé, porteurs de nouvelles mutations proches du site de clivage de la protéine Spike, et jouant un rôle dans l’infectivité virale (Figure 2).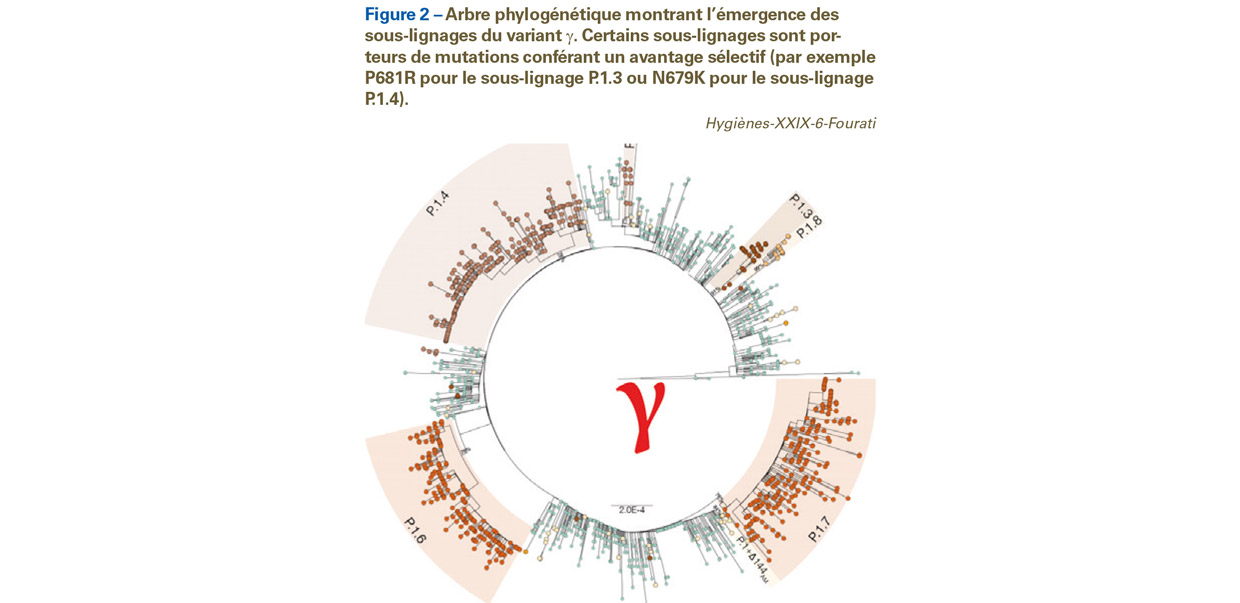
Caractéristiques virologiques et épidémiologiques des principaux variants
Santé publique France classe les variants du SARS-CoV-2 en trois catégories : les variants préoccupants (ou VOC pour variants of concern), les variants à suivre (ou VOI pour variants of interest) et les variants en cours d’évaluation (ou VUM pour variants under monitoring). En novembre 2021, le VOC δ représente la grande majorité des prélèvements séquencés. Le variant Omicron progresse actuellement en France et en Europe.
Variant VOC α (B.1.1.7)
Le variant α (20I/501Y.V1, B.1.1.7, également appelé variant « britannique »), signalé pour la première fois en Grande-Bretagne, a connu une expansion dès décembre 2020 en France et est devenu majoritaire quelques mois plus tard. Il est resté prédominant jusqu’en juin 2021, mais a ensuite été rapidement remplacé par le variant δ. En effet, le VOC α circule très rarement (moins d’1% des variants circulants dès septembre 2021). Sur le plan virologique, le variant α est caractérisé par un profil mutationnel particulier sur la protéine Spike, avec notamment la présence des mutations N501Y, A570D et P681H, ainsi que des délétions en positions 69-70 et 144 de cette protéine (Figures 1 et 3). La substitution N501Y (substitution de l’acide aminé N, asparagine, par le Y, tryptophane, à la position 501) est associée à une augmentation de la transmissibilité par augmentation de l’affinité du RBD de la glycoprotéine Spike pour le récepteur cible ACE2. Sous pression de sélection, de nouvelles mutations ont continué à être sélectionnées sur ce variant, comme l’acquisition de substitutions en position 484 (acquisition d’une lysine E484K ou d’une glycine E484Q). Une étude in vitro récente a montré un impact négatif modéré de la mutation E484K sur la réponse neutralisante post-vaccinale en comparaison avec un variant α sans E484K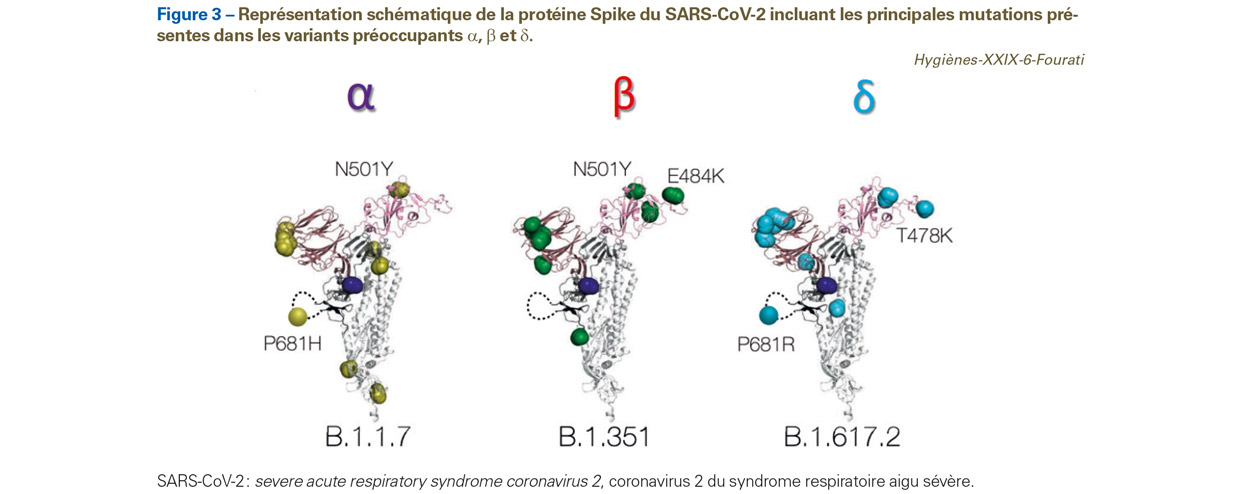
Variants VOC V2 (β) et V3 (γ)
Le VOC β (ou 501Y.V2, lignage B.1.351, également appelé variant « sud-africain ») a été initialement détecté en Afrique du Sud en décembre 2020. Il a ensuite diffusé sur l’ensemble des continents. En France, sa prévalence est en régression également depuis le mois de juillet (il n’est actuellement plus détecté en métropole). Le VOC γ (ou 501Y.V3, lignage P1, également appelé variant « brésilien ») a émergé en décembre 2020 dans la région brésilienne de Manaus (Amazonie). Sa diffusion mondiale a également été rapide au cours du premier trimestre 2021. En France métropolitaine, sa prévalence reste également très faible. Sur le plan virologique, les deux variants β et γ sont porteurs de substitutions aux mêmes positions d’acides aminés dans le RBD (K417N (β), K417T (γ), E484K et N501Y). Les mutations en K417N/T et E484K sont associées à une diminution de la sensibilité à la réponse neutralisante post-vaccinale (Figures 3 et 4). Néanmoins, l’impact est différent entre les deux VOC β et γ, avec une réduction plus importante de l’efficacité des vaccins vis-à-vis du variant β que du variant γ, principalement pour les vaccins à vecteur adénoviral (AstraZeneca14 et Janssen15). Ces observations suggèrent que les substitutions en position 417 et 484 ne suffisent pas à elles seules pour induire une réduction significative de la sensibilité à la réponse neutralisante post-vaccinale. Le NTD de Spike comprend un supersite antigénique ciblé par des anticorps neutralisants. Des délétions au niveau de cette région (240-246), portées par le variant β, semblent faire disparaître des épitopes ciblés par plusieurs anticorps neutralisants.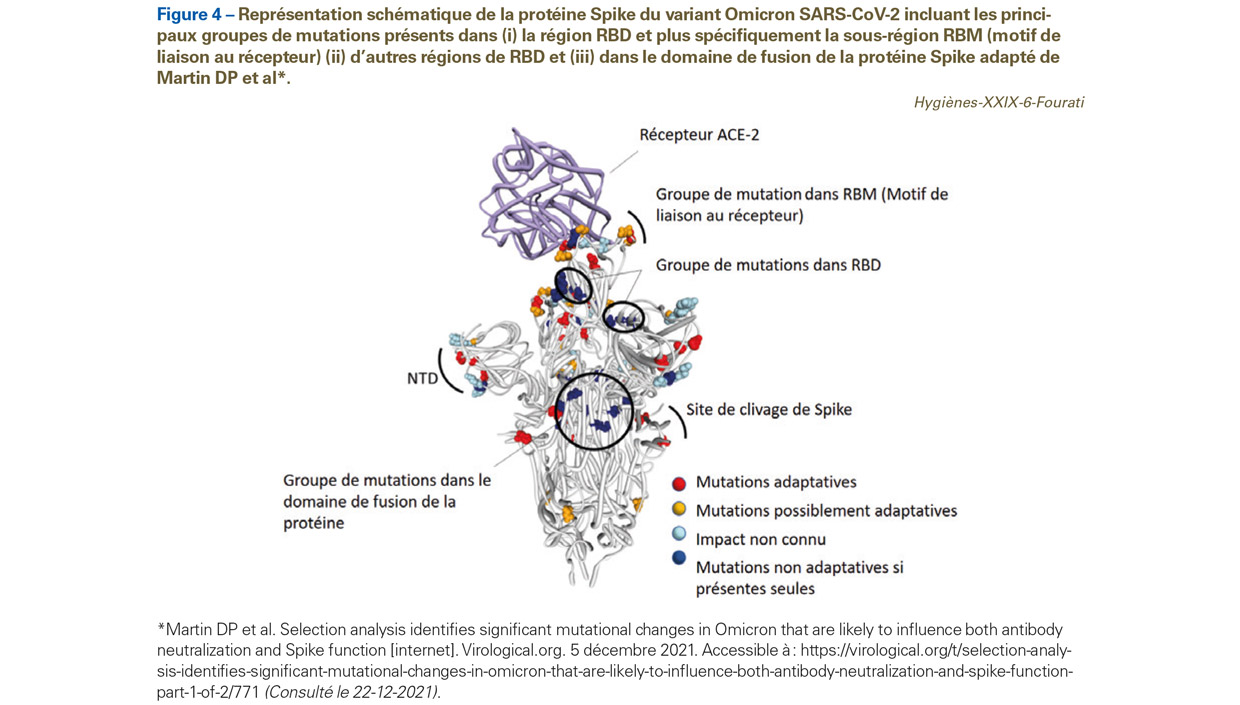
Variant VOC δ
Le variant δ (21A, lignage B.1.617.2, également appelé variant « indien ») a été détecté pour la première fois en Inde occidentale en octobre 2020. Sa diffusion internationale est aujourd’hui largement supérieure à celle des deux autres sous-lignages de variants indiens B.1.617 (B.1.617.1 ou variants et B.1.617.3). Il représente plus de 99% des séquences déposées dans Gisaid16 ayant une date de prélèvement entre le1er octobre 2021 et le 31 octobre 2021. En France, le variant δ domine également, puisqu’il représentait plus de 99% des variants circulants en novembre 2021. Ce variant est progressivement remplacé par le variant Omicron à partir du mois de décembre 2021. La diffusion rapide du Delta à l’échelle planétaire s’explique notamment par une plus grande transmissibilité, avec une augmentation substantielle de son R017 par rapport aux autres variants du SARS-CoV-2 (environ 40 à 60% plus transmissible que le variant α). La plus grande transmissibilité du variant δ est multifactorielle et s’expliquerait par : une augmentation du niveau de réplication virale nasopharyngée comparativement aux variants plus anciens [1] ou une durée d’incubation et un intervalle de transmission interindividuelle plus courts. Des études préliminaires ont suggéré une augmentation du risque d’hospitalisation et de formes sévères en lien avec le VOC δ par rapport aux autres VOC, mais de nombreux facteurs confondants épidémiologiques rendent ces conclusions fragiles [2]. Le variant δ semble également moins sensible à l’action des anticorps neutralisants induits par la vaccination que le VOC α. L’efficacité des vaccins à ARN messager Pfizer18 et Moderna demeure néanmoins élevée contre les formes symptomatiques de Covid-1919, et très élevée contre les formes graves, bien qu’une diminution de la protection au fil du temps ait été observée. Une étude israélienne portant sur la cinétique des anticorps anti-S du SARS-CoV-2 après vaccination ou infection naturelle montre que le titre d’anticorps diminue plus rapidement après six mois chez les sujets vaccinés que chez les sujets précédemment infectés [3]. La reprise à l’automne de l’épidémie dans différentes régions du monde pourrait être liée à la plus forte infectiosité du variant δ, mais aussi à une diminution des niveaux d’anticorps anti-S au cours du temps. Sur le plan virologique, le variant δ est caractérisé par une capacité accrue de propagation dans les cellules respiratoires humaines. Plusieurs hypothèses ont été avancées pour l’expliquer, comme : l’effet de certaines mutations sur une augmentation de l’affinité du virus pour le récepteur ACE-2 (L452R et T478K) (Figure 3) ; in vitro, des pseudovirus exprimant la protéine Spike du variant δ pénètrent dans les cellules cibles exprimant ACE2 beaucoup plus rapidement que celles exprimant les protéines d’autres variants [4,5] ; la substitution P681R (Figure 3) en amont du site de clivage de la furine qui modifie la capacité fusogène de la protéine Spike et augmente l’effet cytopathique induit par ce virus, ce qui favoriserait à son tour une diffusion plus large du virus dans le tissu respiratoire infecté [6] ; ou, enfin, des modifications structurales de l’ARN polymérase du variant δ qui pourraient participer à l’augmentation du niveau de réplication virale. Par ailleurs, la mutation L452R et certaines délétions dans le NTD de Spike (157-158del) pourraient diminuer la sensibilité du virus à certains anticorps neutralisants et de ce fait contribuer à la diminution de l’efficacité vaccinale contre ce variant [7]. En plus des mutations qui caractérisent ce variant, des acquisitions indépendantes de nouvelles mutations sont détectées, comme les substitutions d’intérêt E484A/Q/K, Q613H ou Q677H. Par ailleurs, le sous-lignage de δ AY.4.2 (caractérisé par les substitutions T145H et A222V dans Spike) est suivi de près car voit sa fréquence augmenter au Royaume-Uni (entre 5 et 10% des prélèvements séquencés en octobre 2021). Ce sous-lignage reste aujourd’hui relativement rare en France et dans les autres pays européens. Il serait associé à un taux de croissance plus élevé comparativement aux autres sous-lignages de δ mais ses autres caractéristiques (en termes de sévérité et d’efficacité vaccinale) leur restent similaires.
Variant VOC Omicron
Le variant Omicron (21K, lignage B.1.1.529) a été séquencé pour la première fois le 23 novembre 2021. La majorité des cas a été détectée d’abord en Afrique du Sud mais sa diffusion internationale est rapide et plus de 40 pays ont rapporté la circulation de ce variant sur leur territoire. En France, à la date du 23 décembre 2021, plusieurs centaines de cas d’Omicron ont été identifiées par séquençage. En île de France, il remplace actuellement le variant δ.
Ce variant possède un nombre très important de substitutions (une trentaine), délétions et insertions dans la protéine Spike (Figure 4). Plusieurs substitutions sont localisées au niveau du RBD (domaine de fixation au récepteur notamment, K417N, N440K, G446S, S477N, T478K et E484A), ainsi que des substitutions (Y144F, Y145D), délétions (69-70 et 143-145) et insertion (ins214EPE) dans le domaine NTD de la protéine Spike ; ces mutations sont susceptibles de diminuer fortement la capacité de neutralisation des anticorps post-infection et post-vaccination. Ainsi, plusieurs études préliminaires suggèrent que la capacité neutralisante de sérum de sujets vaccinés par deux doses de vaccin à ARNm (15 jours à 3 semaines après la deuxième dose) est très réduite comparativement aux autres variants préoccupants. En revanche, trois doses vaccinales (vaccins à ARNm/vaccination hétérologue) ou deux doses vaccinales chez un sujet convalescent confèrent un titre d’anticorps neutralisant élevé, qui serait suffisant pour neutraliser l’infection par Omicron (Figure 5). Par ailleurs, la majorité des épitopes ciblés par les lymphocytes T CD8 cytotoxiques ne semblent pas être modifiés pour le variant Omicron (comparativement aux variants d’origine), un élément rassurant confortant les données épidémio-cliniques préliminaires indiquant que la vaccination et/ou les antécédents d’infection naturelle protègent contre les formes graves de l’infection.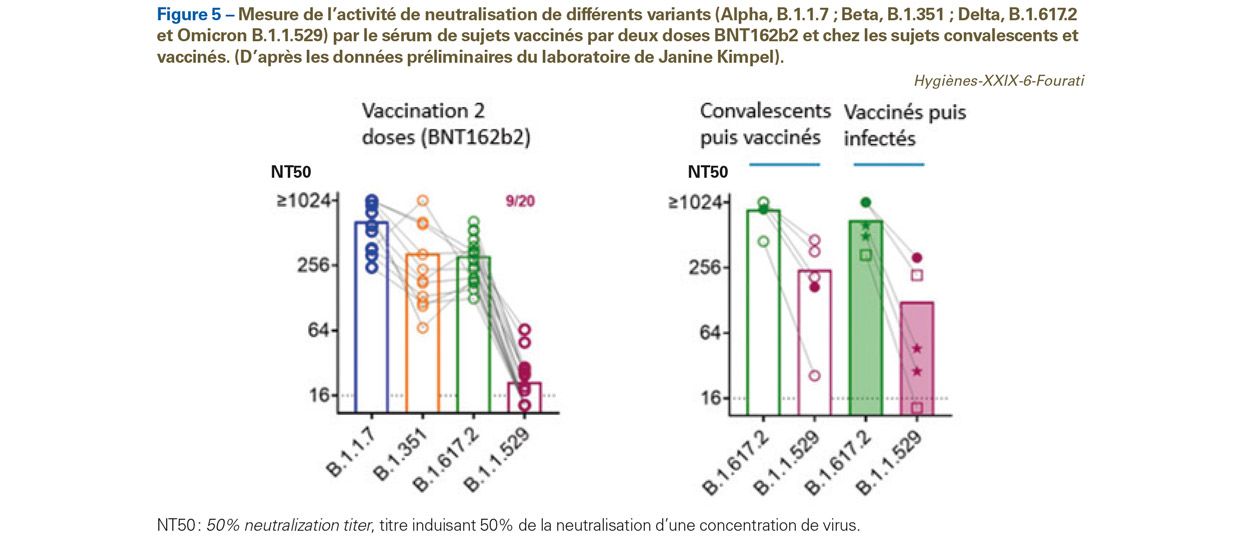
Variants d’intérêt et variants en cours d’évaluation
Les variants d’intérêts (VOI) sont caractérisés par la présence de mutations qui conduisent à des changements d’acides aminés associés à des changements phénotypiques (par exemple, réduction de la sensibilité aux anticorps neutralisants) et ayant un impact prouvé sur la diffusion du variant. Les variants en cours d’évaluation (VUM) sont porteurs de mutations d’intérêt, sans impact prouvé à ce jour sur la santé publique, du moins en France. Au 3 novembre 2021, deux variants étaient classés comme VOI et quatre comme VUM.
Parmi eux, certains font l’objet d’une surveillance particulière. Le VUM B.1.640 a été détecté pour la première fois en République du Congo fin septembre 2021 et circule actuellement en France (<0,5%). Ce variant a été détecté dans plusieurs régions en France (en particulier en Île-de-France et en Bretagne). Avant l’arrivée de l’Omicron, ce variant était le premier à circuler à bas bruit depuis l’arrivée du variant δ, indiquant une potentielle compétitivité vis-à-vis de ce dernier. Ce variant porte plusieurs mutations dans la protéine Spike dont les mutations R346S, N394S, Y449N, F490R, N501Y, et P681H ainsi qu’une délétion de 9 acides aminés dans la protéine S (137-145Del). L’impact spécifique des mutations caractérisant B.1.640 sur ses propriétés fonctionnelles est en cours d’investigation.
Le VOI 21G (C.37, ou variant λ), apparu au Pérou fin 2020, a diffusé en Amérique du Sud. Il est caractérisé par la présence de plusieurs mutations d’échappement à la réponse neutralisante post-vaccinale (substitutions L452Q et F490S et délétion 247-253 dans la protéine Spike). Sa détection reste néanmoins très sporadique en France. Le VOI 21H (B.1.621, ou variant µ) a été responsable de flambées épidémiques d’ampleur cet été en Colombie et en Équateur. Il a été sporadiquement détecté en France et dans d’autres pays européens (Espagne, Italie, Suisse et Royaume-Uni) et fait l’objet d’une surveillance attentive en raison de son profil de mutation (E484K, N501Y, P681H et D950N). Des données phénotypiques préliminaires en provenance du Royaume-Uni suggèrent des propriétés d’évasion immunitaire similaires à celles du VOC β.
Conclusion
Depuis la fin de l’année 2019, le SARS-CoV-2 s’est rapidement propagé dans le monde. Il a évolué en sélectionnant un nombre important de mutations génétiques qui ont facilité sa diffusion en améliorant ses capacités par rapport au virus d’origine (souche Wuhan). Le variant δ, particulièrement contagieux, a été responsable pendant l’été 2021 d’une propagation virale massive à travers le globe. La circulation continue du SARS-CoV-2 dans le monde, en particulier dans les pays où la vaccination n’est pas suffisamment étendue, a favorisé l’émergence d’un nouveau variant préoccupant : Omicron, dont la diffusion très rapide à l’échelle internationale fait craindre une transmissibilité accrue et/ou un échappement immunitaire plus important que les autres VOC dont le variant δ. Au-delà du bénéfice individuel et collectif de la vaccination, celle-ci agit comme une arme limitant l’adaptation génétique virale. Une vaccination massive à l’échelle mondiale crée un environnement hostile à la dynamique de diversification du SARS-CoV-2.
Notes : 1- Le sujet a également fait l’objet d’une mise au point publiée dans la lettre de l’infectiologue : Fourati S, Pawlotsky JM. Actualités sur les variants du SARS-CoV-2. La lettre de l’infectiologue 2021;6. 2- Severe acute respiratory syndrome coronavirus 2, coronavirus 2 du syndrome respiratoire aigu sévère. 3- Perte d’un fragment plus ou moins important d’ADN [acide désoxyribonucléique], constituant une cause de mutation (Le Robert). 4- Créée le 1er janvier 2021, l’ANRS Maladies infectieuses émergentes est la nouvelle agence autonome de l’Institut national de la santé et de la recherche médicale (Inserm), issue de la fusion du consortium français Reacting (Research and action targeting emerging infectious diseases, recherche et action ciblant les maladies infectieuses émergentes) et de l’Agence nationale de recherches sur le sida (ANRS). 5- Initiative mondiale de partage des données sur la grippe aviaire. 6- Acide ribonucléique. 7- Angiotensin-converting enzyme. 8- Transmembrane protease serine 2. 9- Adj. Biologie : qui se juxtapose sans que l’un domine l’autre, en parlant des caractères héréditaires (Le Robert). 10- NSP : nonstructural protein, protéine non structurale. 11- Adenosine deaminase acting on RNA, adénosine-désaminase agissant sur l’ARN. 12- Soit substitution de D (asparagine) par G (glycine) à la position 614 ; dans le cas, plus bas, de P681H/R, substitution de P (proline) par H (histidine) ou R (arginine). 13- N-terminal domain. 14- Cambridge, Royaume-Uni. 15- Beerse, Belgique. 16- https://www.gisaid.org (Consulté le 22-07-2020). 17- Taux de reproduction initial. 18- New York, État de New York, États-Unis. 19- Coronavirus disease 2019, maladie à coronavirus 2019.