Introduction
Candida auris est une levure pathogène phylogénétiquement proche du complexe d’espèce Candida haemulonii et identifiée pour la première fois en 2009 au Japon dans le conduit auditif d’un patient infecté, ce qui lui a valu son nom [1]. Cette levure, dont la niche écologique reste inconnue, est à l’origine de colonisations cutanées chez l’Homme. À noter que C. auris a été récemment isolée de l’environnement (Îles Andaman en Inde, dans des zones marécageuses salines) [2]. Comme les autres espèces du genre Candida, C. auris peut être à l’origine d’infections nosocomiales et invasives, observées notamment chez les patients hospitalisés en service de soins intensifs ou traités par chimiothérapie intensive. C. auris présente des caractéristiques inhabituelles par rapport aux autres Candida sp., comme une acquisition rapide de résistance à divers antifongiques, dont le fluconazole et les échinocandines, ainsi qu’une capacité à persister durablement dans différents environnements, sur du matériel inerte en particulier ; en effet C. auris est doué de thermotolérance et halotolérance, c’est-à-dire que cette espèce est capable de s’adapter à de fortes températures et de fortes concentrations en sel. Ces dernières propriétés lui permettent de disséminer facilement en milieu hospitalier et d’être à l’origine d’épidémies nosocomiales. Ainsi, après son premier isolement en 2009 [1], 68 épidémies à Candida auris ont été signalées à travers le monde avant 2021 [3], avec une émergence simultanée sur tous les continents décrite dans 45 pays [3,4]. La diversité de cette espèce a pu être montrée grâce au séquençage du génome entier qui a conduit à l’identification de clades génétiques répartis dans des zones géographiques spécifiques. À ce jour, il existe six clades de C. auris : clade I d’Asie du Sud, clade II d’Asie de l’Est, clade III d’Afrique, clade IV d’Amérique du Sud, clade V d’Iran [5] et clade VI à Singapour [6]. La grande différence génomique entre les clades a été attribuée à une hypothétique pression sélective exercée par les conditions environnementales spécifiques des lieux géographiques d’émergence de chaque clade. Cette diffusion mondiale et rapide a incité les Centers for Disease Control and Prevention1 (CDC) et le European Centre for Disease Prevention and Control (ECDC) à déclarer C. auris comme une menace sanitaire urgente [7,8]. De même, l’Organisation mondiale de la santé a récemment classé C. auris parmi les agents pathogènes fongiques prioritaires [9]. La prévention de la colonisation à C. auris et la surveillance des patients à risque d’être colonisés ou infectés sont donc des points essentiels. Cette prise en charge doit être précoce et multidisciplinaire. Elle met en jeu les services cliniques qui identifient les patients à risque, les équipes opérationnelles d’hygiène qui coordonnent la surveillance, la prévention et la gestion des épidémies, et les laboratoires de mycologie/microbiologie qui doivent mettre en place des outils diagnostiques efficaces et rapides pour identifier C. auris.
Surveillance en France
En France, la surveillance des infections et des colonisations à Candida auris repose sur plusieurs systèmes interconnectés :
- la déclaration des cas d’infection ou de colonisation par les hygiénistes des hôpitaux à Santé publique France via l’outil e-SIN2. En effet, par leur caractère émergent et devant leur potentielle gravité, les infections nosocomiales à C. auris correspondent aux critères de signalement aux centres d’appui pour la prévention des infections associées aux soins (CPias), aux agences régionales de santé et à Santé publique France, prévus par le décret n° 2017-129 du 3 février 2017 relatif à la prévention des infections associées aux soins ;
- le signalement des cas d’infection ou de colonisation par les laboratoires de mycologie/microbiologie de centre hospitalier ou centre hospitalier universitaire auprès du Centre national de référence des mycoses invasives et antifongiques (CNRMA) associé à l’envoi des souches selon les recommandations du Haut Conseil de la santé publique (HCSP) du 14 juin 2019 et de la note publiée le 17 avril 20233, permettant la détermination de la sensibilité aux antifongiques par la méthode standardisée du European Committee on Antimicrobial Susceptibility Testing (EUCAST), ainsi qu’une analyse épidémiologique en combinant les données de génome entier et les données cliniques afin d’avoir un suivi de l’émergence de l’espèce et des éventuelles transmissions.
Épidémiologie française : données disponibles au 17 avril 2023
Pour ces données, les cas sont définis de façon consensuelle par les patients détectés porteurs en culture et éventuellement infectés au niveau d’un prélèvement superficiel ou profond. Le premier cas européen a été observé en France en 2007 de façon rétrospective chez un patient qui avait été précédemment hospitalisé en Inde [10]. Le premier cas rapporté en France de façon prospective a été diagnostiqué en 2017 chez un patient ayant voyagé en Inde et en Iran avec des prélèvements positifs retrouvés dans la gorge, les urines et le liquide péritonéal [11]. Grâce aux différents canaux cités ci-dessus, un total de 10 cas d’infection, dont la moitié était isolée d’hémocultures, et 14 cas de colonisation à C. auris ont été recensés depuis 2007 dans 14 hôpitaux sur tout le territoire français (Figure 1). Parmi tous les patients détectés, la grande majorité avaient séjourné dans différents pays d’endémicité connue (Inde, Pakistan, Dubaï, Koweït, Madagascar, Kenya, Russie, Niger, Espagne, Égypte). Parmi tous ces cas, seuls deux épisodes de transmission entre patients ont été avérés (un épisode avec un cas de colonisation secondaire en 2021 [12] et un avec deux cas de colonisation secondaire en 2022). Pour chacun de ces cas, des mesures de prévention de la transmission ad hoc ont été prises, conformément aux recommandations 2019 du HCSP. Les cas d’infection et de colonisation à C. auris recensés au CNRMA en France sont rapportés au niveau européen via le groupe de surveillance européen (Candida auris Survey Collaborative Group) mis en place et géré par l’ECDC [13].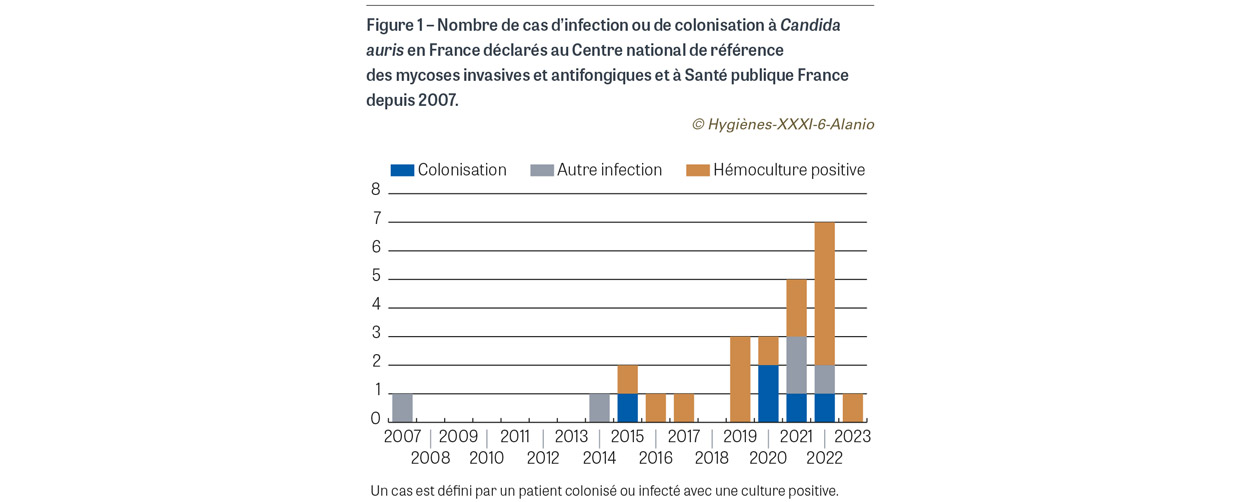
Diagnostic, outils et stratégies diagnostiques
Méthodes conventionnelles
Les outils diagnostiques conventionnels utilisés en routine dans les laboratoires de mycologie/microbiologie permettent l’identification de Candida auris. Différentes stratégies peuvent être mises en place pour améliorer sa mise en évidence. Il faut avoir à l’esprit que C. auris colonise la peau, gîte de plusieurs levures comme C. parapsilosis, ainsi que le milieu hospitalier. C. auris peut donc être présente en culture avec d’autres espèces, rendant parfois difficile son isolement et son identification au regard notamment de son délai de culture (cf. infra). Les techniques de coloration et la visualisation microscopique de cette levure à partir d’un prélèvement clinique ou à l’état frais d’une culture ne sont pas différentes de celles utilisées pour les autres levures du genre Candida. En effet, C. auris se colore parfaitement grâce aux colorations courantes de microbiologie (calcofluor-white, coloration de Gram, bleu de lactophénol…) et apparaît comme une levure bourgeonnante, ovoïde à allongée, d’une taille de 2 à 3 µm sur 2,5 à 5 μm [1], avec des pseudo-filaments rares. Candida auris se cultive sur les milieux de culture standards de bactériologie (gélose au sang ou gélose chocolat) ainsi que sur les milieux de mycologie (Sabouraud ou milieux chromogènes) qui permettent l’isolement et la différenciation des principales espèces de Candida. À noter que l’utilisation de bouillons d’enrichissement (Sabouraud liquide et NaCl 10%) permet d’améliorer la sensibilité de la culture [14]. En cas de suspicion ou de recherche de colonisation à C. auris chez un patient, il est recommandé d’incuber les cultures pendant au moins 10 jours, idéalement à 40°C ou sinon à 37°C, ce qui favorise la croissance de C. auris par rapport aux autres espèces. En 48 heures à 35°C, les colonies de C. auris apparaissent crème pâle à bleu clair sur CandiSelectTM (Bio-Rad Laboratories, Hercules, CA, États-Unis) (Figure 2A) ou beige à rose avec des bordures pâles sur ChromaticTM Candida (Liofilchem, Roseto degli Abruzzi, Italie) ou CHROMagarTM Candida (CHROMagar, Paris, France) (Figure 2B-C) [15]. Avec ces milieux, il n’est pas possible de distinguer C. auris d’autres espèces pathogènes apparentées du complexe C. haemulonii ou même de C. parapsilosis parce que les colonies de C. auris présentent un phénotype (couleur et forme de la colonie) similaire. Dans ce contexte, deux nouvelles formulations de milieu chromogène ont été commercialisées, le CHROMagarTM Candida Plus (CHROMagar) et le HiCromeTM MDR C. auris Selective Agar Base (HAMA) (HiMedia Laboratories, Einhausen, Allemagne). Ce dernier milieu chromogène contient un mélange d’antifongiques qui le rend sélectif pour les levures multirésistantes. En 48 heures, les colonies de C. auris apparaissent roses à violettes sur HAMA ou bleu claires avec un halo bleu sur CHROMagarTM Candida Plus (Figure 2D-E). L’évaluation des cinq milieux chromogènes disponibles dans le commerce pour l’identification présomptive de C. auris a été faite dans la littérature, et il s’avère que la gélose CHROMagarTM Candida Plus est la plus performante pour son isolement, bien que de lecture difficile [15]. Par ailleurs, afin de permettre la sélection de C. auris lors de mélange avec différentes espèces de Candida sur des boîtes de culture, une gélose CHROMagarTM Candida supplémentée avec 32 mg/L de fluconazole a été mise au point [16]. Ainsi, la pousse des souches de Candida sensibles au fluconazole est inhibée et seuls les isolats de C. auris résistants au fluconazole sont sélectionnés. L’utilisation de ce milieu n’est pas recommandée en France en raison notamment de l’existence de souches de C. auris sensibles au fluconazole appartenant au clade II principalement.
Moyens des laboratoires
Les colonies suspectes de Candida auris doivent être identifiées de façon fiable et rapide par différents systèmes d’identification en fonction du matériel disponible dans les laboratoires de mycologie/microbiologie. Certains utilisent encore les tests phénotypiques biochimiques pour identifier les bactéries et les champignons en culture. Dans ce cas, l’identification de C. auris ne peut pas se faire correctement car ces schémas d’assimilation et de fermentation des sucres sont similaires à ceux d’autres espèces de levures étroitement apparentées. De plus, certains systèmes d’identification procèdent à une identification des espèces sur la base de la correspondance la plus proche, plutôt que sur la base d’une correspondance parfaite. Ainsi, les systèmes API 20 C AUX, API ID 32 C, ou Vitek 2-YST (bioMérieux, Marcy-l’Étoile, France) peuvent identifier à tort C. auris comme Rhodotorula glutinis, Candida sake ou C. haemulonii [17]. D’autres laboratoires sont équipés d’un spectromètre de masse MALDI-TOF MS4 pour l’identification des micro-organismes. Les premières identifications de C. auris par MALDI-TOF MS n’ont pas été possibles parce que cette nouvelle espèce n’était pas implémentée dans les bases de données. Depuis, les fabricants l’ont ajoutée à leurs bases de données, permettant l’identification rapide et correcte de tous les clades de C. auris [18,19]. Il est néanmoins important de contrôler que les bases de données utilisées dans les laboratoires de mycologie/microbiologie sont à jour et capables d’identifier C. auris. Pour aider les laboratoires, le CDC a créé un algorithme qui détaille les étapes nécessaires à suivre et à vérifier afin d’améliorer l’identification de C. auris par les méthodes phénotypiques conventionnelles, en fonction des systèmes utilisés5. De plus, la Société française de mycologie médicale (SFMM) et le CNRMA recommandent d’utiliser une souche de C. auris de collection comme contrôle qualité afin de vérifier les différents systèmes d’identification des levures.
Techniques de diagnostic moléculaire
En plus des méthodes de diagnostic mycologiques conventionnelles, des techniques de diagnostic moléculaire ont récemment vu le jour pour identifier rapidement et spécifiquement C. auris. Avant l’étape d’amplification, l’étape d’extraction d’ADN6 à partir de prélèvement clinique ou d’une culture, critique en mycologie en raison de l’épaisseur de la paroi des champignons, ne doit pas être négligée. À partir d’ADN extrait de colonies pures, l’identification basée sur le séquençage de la région internal transcribed spacer (ITS) et sur les régions D1/D2 du gène ribosomique, permet de différencier C. auris d’autres espèces étroitement apparentées [20]. Cependant, le séquençage n’est pas possible à partir d’échantillons cliniques qui sont le plus souvent plurimicrobiens (hors biopsies profondes et liquides stériles). Un certain nombre de kits commerciaux de test PCR7 en temps réel ont été développés, basés sur différentes techniques, en utilisant des amorces ou des thermocycleurs différents (Tableau I) [21,22,23]. En plus des kits commerciaux « prêt à l’emploi », certains laboratoires ont aussi développé des PCR quantitatives « maison » basées sur la technologie TaqMan (Thermo Fisher Scientific, Waltham, MA, États-Unis) et ciblant la région ITS de C. auris qui sont aisément utilisées en diagnostic et en criblage [12,23,24]. Ces méthodes moléculaires ont l’avantage d’être effectuées directement sur le prélèvement clinique (écouvillonnage ou prélèvement plus profond), permettant de s’affranchir de l’étape de culture et de rendre un résultat très rapidement (<24 h au mieux). Du fait de leur très bonne sensibilité, ces techniques peuvent « rattraper » les résultats des cultures à charge fongique faible, qui pourraient être rendus négatifs à tort. Ces outils de PCR en temps réel étant très spécifiques de C. auris, ils peuvent également être utilisés pour analyser une colonie suspecte après extraction d’ADN.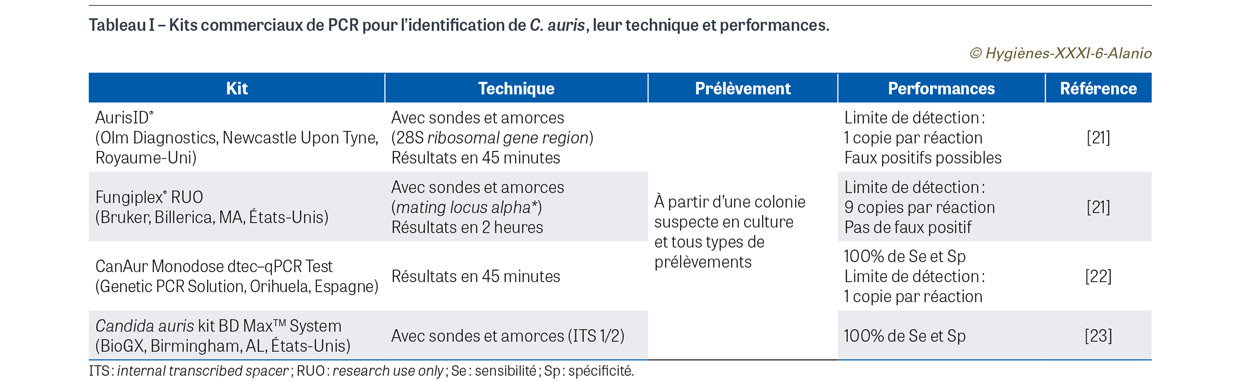
Complémentarité des techniques
Ces PCR sont donc des outils complémentaires utiles à la culture du prélèvement. Cependant, l’interprétation peut être difficile lorsqu’un prélèvement est positif en PCR et négatif en culture. Dans ce cas, le patient doit être impérativement prélevé sur plusieurs sites (aisselles, aines, mains, pieds, nez, selles, etc.) avec réalisation de cultures afin de maximiser les chances d’isolement positif. Plusieurs combinaisons de résultats sont donc possibles et l’interprétation est alors la suivante8 :
- si culture positive : cas certain ;
- si PCR positive avec culture négative : cas possible, renouveler et élargir les sites de prélèvement. Si au moins 2 PCR positives : cas possible. Si 1 seule PCR positive suivie d’au moins 4 PCR et cultures négatives à une semaine d’intervalle : pas de portage.
La recherche d’une colonisation à C. auris chez les patients à risque s’effectue par écouvillonnage de sites cutanés bien définis. Il est recommandé d’effectuer un prélèvement par écouvillon au niveau des plis axillaires et inguinaux, et d’ajouter un prélèvement nasal qui permet d’augmenter la sensibilité du dépistage de 60% à 80% [25]. Plus le nombre de sites cutanés prélevés est important, plus la sensibilité augmente ; ainsi, la combinaison narines, paumes des mains, extrémités des doigts et orteils, zone périanale et pli inguinal permet une sensibilité de 97,8% [25]. Concernant les stratégies diagnostiques de colonisation à C. auris, le CNRMA rappelle dans sa note du 17 avril 20239 que le dépistage par culture d’écouvillons inguinal, axillaire et nasal est préconisé pour tout patient :
- hospitalisé dans les douze mois précédents, notamment s’il a été rapatrié d’une réanimation d’un pays étranger ; dépistage à réitérer si réadmission dans les douze mois suivant le retour ;
- antérieurement colonisé ou infecté par C. auris ;
- contact d’un cas.
Enfin, comme pour toute autre suspicion d’infection fongique invasive causée par Candida spp., le dosage de marqueurs fongiques peut être utilisé comme outil diagnostique. Le 1,3-β-D-glucan (BDG), composant de la paroi fongique, est à ce titre considéré comme un marqueur « pan-fongique ». Sa quantification dans le sérum est recommandée lors d’une suspicion de candidose invasive en raison de la faible sensibilité des hémocultures et du délai de rendu des hémocultures positives [26]. Les causes de fausse positivité du dosage de BDG, nombreuses, doivent néanmoins être écartées avant de conclure. Mais il a été montré que le taux de BDG sérique était inférieur chez les patients « fongémiques » à C. auris versus C. albicans [27,28]. De plus, le BDG a été quantifié dans les surnageants de culture de dix souches cliniques de C. auris isolées d’hémocultures et les auteurs ont pu montrer que le niveau de BDG était inférieur à celui retrouvé avec les surnageants de culture de souches de Candida albicans [29].
Génotypage des souches de C. auris
Grâce aux nombreuses études phylogénétiques réalisées depuis les premières épidémies à C. auris, la diversité génétique de l’espèce, son génome et son taux de mutation sont désormais assez bien connus [30,31]. De plus, il existe une méthode de génotypage par microsatellites rapide et bon marché [32]. Le séquençage du génome entier des isolats de C. auris reçus au CNRMA a permis de comparer les isolats entre eux et avec les génomes de référence des différents clades connus. La majorité des isolats retrouvés en France appartiennent au clade I (sud-asiatique/indien), deux isolats seulement appartiennent au clade III (sud-africain). Ces analyses combinées avec les données épidémiologiques ont ainsi pu confirmer les cas de transmission entre patients, les isolats d’un même patient ou d’une même source ayant un nombre de SNP10 communs très faible (<10 sur l’ensemble du génome) [12].
Sensibilité aux antifongiques de C. auris
L’une des caractéristiques les plus alarmantes de C. auris est le pourcentage d’isolats résistants aux antifongiques. Ainsi, dès lors que Candida auris est identifié en culture, un antifongigramme doit être effectué. Cela permet de déterminer les concentrations minimales inhibitrices (CMI) aux différents antifongiques et d’aider les cliniciens dans la prise en charge de leur patient. Les méthodes commerciales par bandelettes Etest® (bioMérieux, Marcy-l’Étoile, France) imprégnées d’un gradient d’antifongique sur gélose RPMI11, utilisées par un très grand nombre de laboratoires de mycologie/microbiologie, montre des résultats comparables à ceux obtenus avec les méthodes de références américaines du Clinical and Laboratory Standards Institute (CLSI) ou européennes (EUCAST) [33]. Cependant, à ce jour, il n’existe aucun clinical breakpoint12 établi par le CLSI ou l’EUCAST pour C. auris, ce qui explique que l’interprétation des CMI soit basée sur les breakpoints épidémiologiques (ECOFF ou ECV13). De nombreux laboratoires se réfèrent donc aux breakpoints provisoires établis par le CDC (Tableau II). Sur la base de ces seuils, une étude indienne a montré qu’environ 25% des souches se sont révélées résistantes à plusieurs classes d’antifongiques, les catégorisant alors comme multi-drug resistant (MDR) [34]. Il est néanmoins à noter que, parmi ces souches, certaines provenaient de la dissémination d’un clone MDR, ce qui peut biaiser ce pourcentage.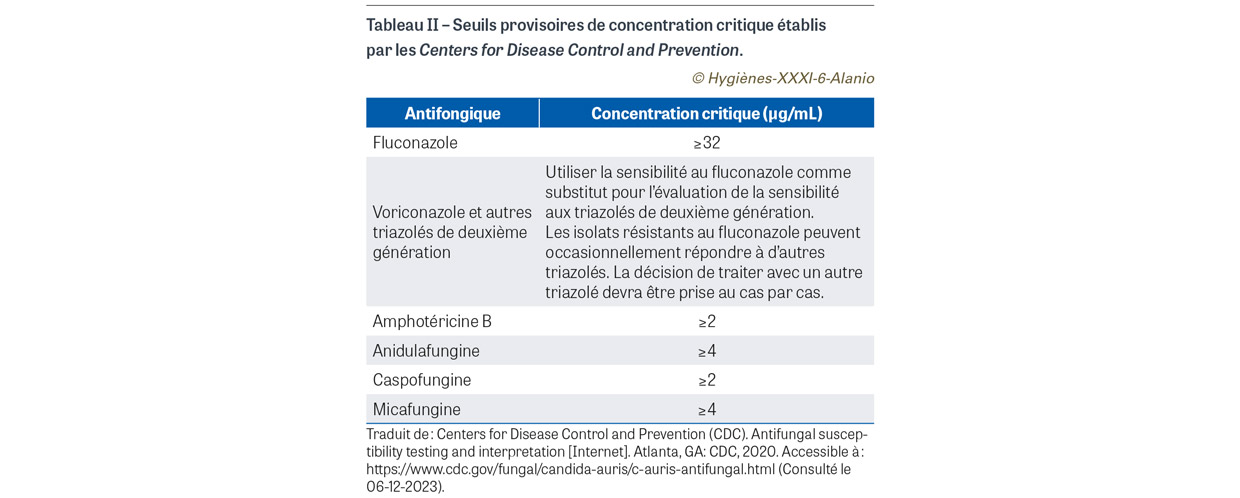
Données françaises de sensibilité aux antifongiques
La majorité des souches de C. auris retrouvées en France ayant été transmises au CNRMA, la détermination de la sensibilité aux antifongiques a pu être établie par la méthode EUCAST (Tableau III). Tous les isolats recensés étaient résistants au fluconazole, six avaient des CMI très élevées à la 5FC14 mais tous étaient sensibles aux échinocandines. Ces profils étaient similaires avec les données de la littérature.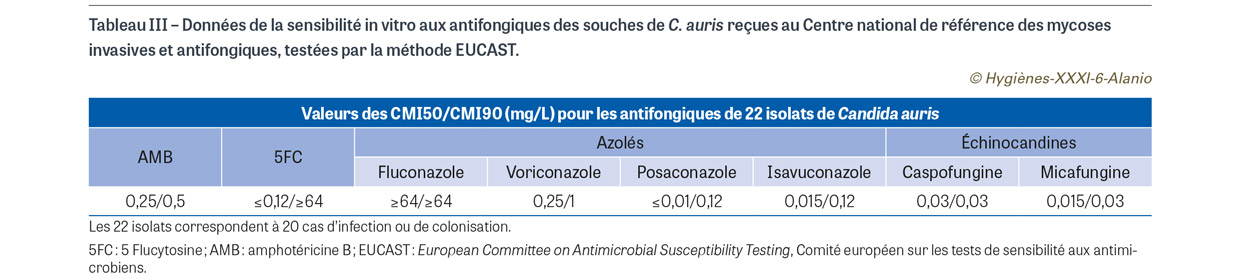
Prévention, désinfection, résistance aux désinfectants, décolonisation
En France, les recommandations nationales de prévention de l’infection à C. auris sont celles publiées en 2019 par le HCSP [35]. Mais la recrudescence mondiale actuelle des cas pourrait amener des révisions de ces recommandations.
Dépistage des cas
Pour contrôler l’infection et limiter son risque de diffusion épidémique, il convient d’identifier le plus précocement possible les cas afin d’établir des mesures de prévention efficaces autour d’eux. La première approche concerne le dépistage systématique des sujets à risque d’être colonisés par C. auris. Le HCSP a exclu en 2019 tout dépistage systématique en dehors des patients au portage antérieur connu et de ceux hospitalisés dans une zone, une région ou un établissement où une épidémie à C. auris est en cours. Toutefois, ce dernier critère s’est avéré inopérant car trop complexe à décliner de façon opérationnelle. Certains établissements particulièrement exposés au risque de C. auris, comme l’Assistance publique-Hôpitaux de Paris ou certaines structures régionales (CPias Nouvelle-Aquitaine) où des patients venant de zones d’endémie peuvent être nombreux, ont proposé des stratégies de dépistage renforcées (voir supra). Au niveau international, plusieurs pays se sont calés sur l’approche connue avec les bactéries hautement résistantes aux antibiotiques émergentes (BHRe) et cette tendance tend à gagner du terrain. Les CDC américains suggèrent de dépister tous les patients ayant séjourné au moins une nuit dans un hôpital hors des États-Unis pendant l’année précédente, et en particulier ceux colonisés par des BHRe car la double colonisation est fréquente. Pour le dépistage, l’approche américaine a été reprise en la limitant aux pays ayant rapporté des cas. La vision française s’est inspirée de la stratégie mise en place mais en élargissant à tous les patients transférés d’un établissement étranger ou d’une unité en France où des cas ont été détectés pendant l’hospitalisation. Le gouvernement britannique, quant à lui, a mis en ligne en juin 2023 une consultation sur son projet de nouvelles recommandations15. En France, le HCSP propose, en cas d’identification fortuite d’un cas, un dépistage exhaustif des contacts en remontant jusqu’à quatre semaines avant la date du premier prélèvement positif. Dans d’autres pays, les approches sont plus restrictives et se limitent par exemple aux voisins de chambre et aux patients dont la charge en soins est la plus élevée. Les services de soins intensifs et les situations de cas secondaires avérés font consensus pour un dépistage systématique de tous les patients contact. Le dépistage du portage chez les professionnels de santé n’est pas recommandé.
Précautions complémentaires
Autour d’un patient porteur de C. auris, les mesures préconisées par le HCSP sont les suivantes :
- hospitaliser le patient dans une chambre individuelle ;
- réaliser un dépistage hebdomadaire ;
- appliquer les précautions complémentaires de type contact (PCC) et dédier du personnel paramédical au patient ; ou appliquer les PCC et la marche en avant en isolant le patient dans l’objectif de réduire le nombre de contacts le jour et la nuit.
En cas de situation épidémique, il convient d’arrêter les transferts dans l’attente de la maîtrise de la situation, de réaliser une cartographie des cas, des contacts et des patients indemnes, et d’attribuer du personnel dédié à chaque secteur en fonction de la situation. Pour la cartographie, il paraît raisonnable de s’assurer au préalable de l’absence de colonisation concomitante d’un cas avec une BHRe qui représenterait une perte de chance pour un autre patient colonisé à C. auris mais indemne de BHRe.
Gestion de l’environnement et désinfection
C. auris a démontré une aptitude à survivre de façon prolongée dans l’environnement et à coloniser rapidement de nombreuses surfaces de la chambre d’un patient porteur. Il est donc nécessaire de renforcer le bionettoyage des chambres de patients colonisés ou infectés à C. auris, et en particulier celui des surfaces fréquemment touchées pour éviter la dissémination du pathogène, en complément de l’hygiène des mains. Les produits répondant aux normes européennes de levuricidie pour la désinfection des surfaces (EN 13624, EN 17387 et EN 16615) utilisent Candida albicans ATCC® 10231TM (American Type Culture Collection, Manassas, VA, États-Unis) comme souche test, mais il est possible d’y ajouter C. auris en souche additionnelle. À ce jour, il n’est pas décrit de comportement différent entre C. albicans et C. auris vis-à-vis des désinfectants Toutefois, les produits oxydants sont souvent préconisés dans les recommandations internationales car de délai d’action plus rapide. Certains pays comme les États-Unis ont des listes de produits agréés pour ce pathogène16, ce qui n’est pas le cas en France. À la sortie de la chambre d’un patient porteur de C. auris, il est nécessaire de garantir au patient qui sera admis ensuite dans cette chambre l’absence de C. auris résiduel dans l’environnement. Le bionettoyage peut être complété par une désinfection chimique (désinfection des surfaces par voie aérienne par diffusion d’une solution de peroxyde d’hydrogène) ou physique (désinfection des surfaces par des rayonnements ultraviolets) réalisée hors présence humaine. Chaque établissement de santé doit définir ses règles dans ce domaine et la façon dont il garantit à l’usager la qualité des processus. Il est important de limiter l’usage de dispositifs médicaux partagés entre patients colonisés et non colonisés et de s’assurer que les responsabilités de nettoyage et de désinfection de l’environnement et des dispositifs médicaux ont été clairement établies, car c’est un écueil souvent rencontré dans les situations épidémiques17.
Décolonisation des porteurs
Le HCSP précisait en 2019 qu’aucune décolonisation des patients n’était recommandée en l’état actuel des connaissances. Cette position reste consensuelle au niveau international. En effet, si la chlorhexidine s’est avérée in vitro efficace à concentration élevée vis-à-vis de C. auris [36], il n’y a pas encore de preuve clinique de l’impact de son usage sur la baisse du portage ou des infections associées à C. auris. En situation épidémique, la décolonisation cutanée s’est avérée efficace chez certains patients [37]. La connaissance du microbiote cutané des patients porteurs de C. auris se développe et sa modélisation aidera dans le futur à mieux déterminer les stratégies de prévention potentiellement efficaces, notamment celle pour la décolonisation [38].
Conclusion
Candida auris est une levure émergente, possédant un potentiel de dissémination fort et un spectre de susceptibilité aux antifongiques pouvant être très réduit. Cette levure initialement sensible peut acquérir rapidement une résistance à un ou plusieurs antifongiques (multirésistance) et disséminer localement ou de façon plus large. En France, les C. auris qui ont été isolées étaient toutes résistantes au fluconazole. Les mesures de contrôle de la diffusion sont d’autant plus efficaces qu’elles sont appliquées tôt, ce qui justifie une identification rapide des cas. Il est primordial que les cliniciens soient sensibilisés au dépistage des patients à risque et que les laboratoires de mycologie/microbiologie aient la capacité d’identifier rapidement les souches suspectes, ce d’autant que les outils disponibles pour la détection et l’identification précise de C. auris se sont considérablement améliorés. Le contrôle de l’épidémie est basé sur une prise en charge multidisciplinaire entre les équipes de prévention du risque infectieux, les cliniciens et les laboratoires de mycologie/microbiologie.
Notes :
1- Centres pour le contrôle et la prévention des maladies.
2- Signalement des infections nosocomiales en ligne.
3- Centre national de référence des mycoses invasives et antifongiques (CNRMA). Note du centre national de référence des mycoses invasives et antifongiques (CNRMA)/LA INuSuAl (Identification numérique surveillance alerte), de la Société française de mycologie médicale (SFMM) et de la Société française d’hygiène hospitalière (SF2H) sur l’épidémiologie et la surveillance des infections à Candida auris en France : mise à jour du 17/04/2023. Paris : Institut Pasteur, 2023. 4 p. Accessible à : https://www.sfm-microbiologie.org/wp-content/uploads/2023/06/Note-auris-Juin-2023.pdf (Consulté le 06-12-2023).
4- Matrix assisted laser desorption ionization time of flight mass spectometry, spectrométrie de masse à temps de vol pour la désorption-ionisation laser assistée par matrice.
5- National Center for Emerging and Zoonotic Infectious Diseases. Algorithm to identify Candida auris based on phenotypic laboratory method and initial species identification. Atlanta, GA: Centers for Disease Control and Prevention, 2019. 11 p. Accessible à : https://www.cdc.gov/fungal/candida-auris/pdf/Testing-algorithm_by-Method_508.pdf (Consulté le 06-12-2023).
6- Acide désoxyribonucléique.
7- Polymerase chain reaction, réaction en chaîne par polymérase.
8- CNRMA, op. cit.
9- Ibid.
10- Single nucleotide polymorphism, polymorphisme d'un seul nucléotide.
11- Roswell Park Memorial Institute.
12- Point (seuil de concentration) critique clinique.
13- Epidemiological cut-off value, valeur de seuil épidémiologique.
14- 5-fluorocytosine ou flucytosine.
15- UK Health Security Agency. Candida auris: update to management guidance [Internet]. London: Government of the United Kingdom, 2023. Accessible à : https://www.gov.uk/government/consultations/candida-auris-update-to-management-guidance (Consulté le 06-12-2023).
16- United States Environmental Protection (EPA). List P: Antimicrobial products registered with EPA for claims against Candida auris [Internet]. Washington, DC: EPA, 2023. Accessible à : https://www.epa.gov/pesticide-registration/list-p-antimicrobial-products-registered-epa-claims-against-candida-auris (Consulté le 06-12-2023).
17- Centers for Disease Control and Prevention (CDC). Infection prevention and control for Candida auris [Internet]. Atlanta, GA: CDC, 2023. Accessible à : https://www.cdc.gov/fungal/candida-auris/c-auris-infection-control.html (Consulté le 06-12-2023).