C’est pourquoi les bactériologistes ont développé des techniques dites « rapides » de détection de la résistance des bactéries aux antibiotiques, que nous allons aborder au cours du sixième volet de cette rubrique. Contrairement aux tests directement réalisables sur le prélèvement clinique, ces techniques nécessitent une culture pure de la souche bactérienne suspectée ; elles ne font donc pas l’économie de la phase dite de « primo-culture » de l’échantillon clinique sur milieux solides, sélectifs ou non, ou sous la forme de flacons positifs d’hémoculture. Avec ces tests rapides, le délai d’obtention des résultats microbiologiques est réduit d’une douzaine d’heures, soit un premier rendu de données de sensibilité aux antibiotiques dès 24 heures (Figure 1). Ces approches sont particulièrement intéressantes les veilles de week-end ou de jour férié, qui retardent de 24 à 48 heures la lecture des antibiogrammes dans les institutions ne disposant pas d’une microbiologie accessible sept jours sur sept. Certains tests, très simples et peu coûteux, peuvent être réalisés dans tous les laboratoires de microbiologie ; d’autres, plus onéreux ou complexes car nécessitant un appareillage dédié, sont disponibles dans des structures plus spécialisées. L’hygiéniste peut bénéficier des informations précoces fournies par ces outils pour émettre ses recommandations ; il doit néanmoins en connaître les limites.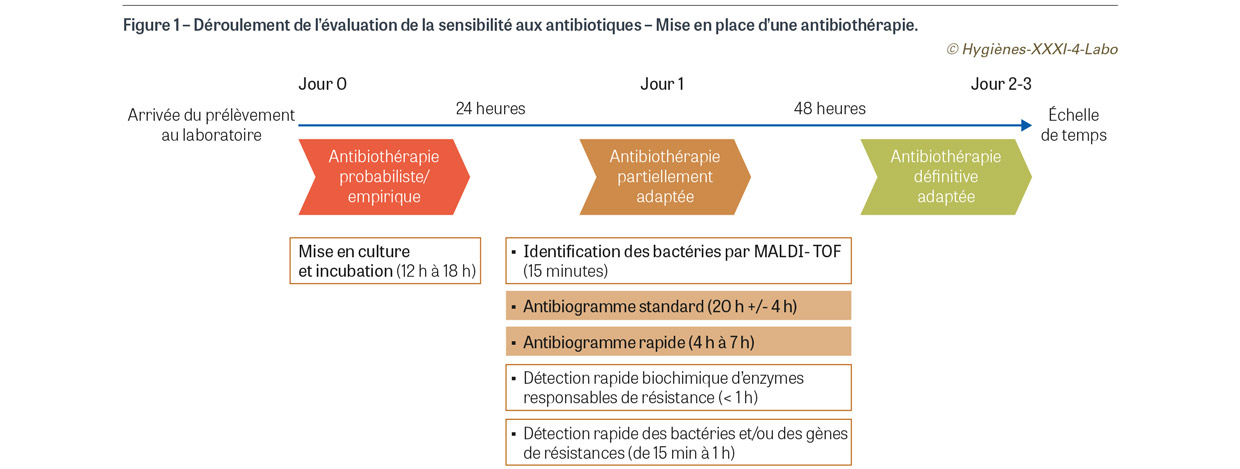
L’antibiogramme rapide : la quête du Graal ?
Lecture anticipée des antibiogrammes standard par diffusion (antibiogramme par la méthode des disques)
Nous avons vu dans le précédent volet de cette rubrique que l’antibiogramme par la méthode de diffusion en milieu gélosé à partir de disques imprégnés d’antibiotique était une méthode historique encore très utilisée par les microbiologistes en France [4]. Pour rappel, la lecture des diamètres d’inhibition est réalisée après une incubation de 20±4 heures ; ces diamètres sont ensuite corrélés à des concentrations minimales inhibitrices au moyen de courbes de concordance [1]. La lecture de ces diamètres est de plus en plus souvent automatisée grâce à des caméras. L’antibiogramme peut non seulement être réalisé à partir de colonies isolées sur une boîte de Pétri gélosée mais aussi directement à partir d’un flacon d’hémoculture positif. Dans ce cas, il est possible de raccourcir la durée d’incubation et de lire les diamètres d’inhibition dès 4, 6 et 8 heures d’incubation [5]. Des diamètres critiques spécifiques ont été établis pour interpréter ces diamètres d’inhibition après un temps de culture raccourci, mais uniquement pour les espèces bactériennes les plus couramment identifiées en médecine humaine : Escherichia coli, Klebsiella pneumoniae, Pseudomonas aeruginosa, Staphylococcus aureus, Streptococcus pneumoniae, Enterococcus faecalis, E. faecium et Acinetobacter baumannii. Cette approche ne nécessite pas d’investissement supplémentaire en consommables de laboratoire. Elle ne concerne cependant que les laboratoires utilisant la méthode de diffusion en milieu gélosé à partir de disques. Le temps gagné n’est parfois que de quelques heures, ce qui ne permet pas toujours d’adapter la prise en charge du malade : si le résultat devient disponible quand le médecin est parti, la prise en charge thérapeutique reste inchangée.
Nouvelles techniques dites antibiogrammes rapides
En parallèle, des industriels ont développé des technologies spécifiques permettant d’estimer plus rapidement les concentrations minimales inhibitrices d’un panel d’antibiotiques à partir d’une hémoculture positive. Les techniques utilisées font appel à des caméras et des détecteurs de nouvelle génération capables de mesurer la croissance bactérienne (qui se traduit par une augmentation de turbidité du milieu) en présence d’un gradient d’antibiotique (c’est-à-dire une gamme de concentration croissante de la molécule d’antibiotique) dans un milieu semi-gélosé et de la comparer à la croissance bactérienne sans antibiotique ou à des courbes de croissance déjà établies avec et sans antibiotique. Les résultats peuvent être obtenus en 4 à 7 heures [6-8]. Ces technologies nécessitent un matériel spécifique ; leur coût unitaire dépasse la centaine d’euros et varie en fonction du nombre de tests réalisés par les laboratoires. De plus elles ne sont applicables aujourd’hui qu’aux cultures positives en flacons d’hémoculture.
Tests rapides de détection des résistances
Contrairement aux tests précédents qui permettent d’obtenir plus rapidement un antibiogramme complet concernant plusieurs familles d’antibiotiques, il existe des tests unitaires qui permettent de rechercher individuellement la présence d’une résistance à une seule classe d’antibiotique : bêtalactamines (céphalosporines de 3e génération, carbapénèmes, bêtalactamines à activité antistaphylococcique), glycopeptides (vancomycine…) ou polymyxine (colistine).
Détection ciblée de résistances acquises aux antibiotiques
La recherche spécifique de marqueurs de résistance acquise est une approche simple et très spécifique. Trois grandes familles de tests sont utilisées selon la cible recherchée : gène (tests moléculaires), protéine (tests antigéniques) ou activité enzymatique (Tableau I). Concernant les deux premières catégories de tests, la principale limite est que l’« on ne trouve que ce que l’on cherche » : un résultat négatif n’exclut pas la présence d’un autre mécanisme ou support génétique n’appartenant pas au panel initial recherché. Néanmoins, lorsque les cibles recherchées sont bien adaptées à l’épidémiologie locale, ces tests sont très performants.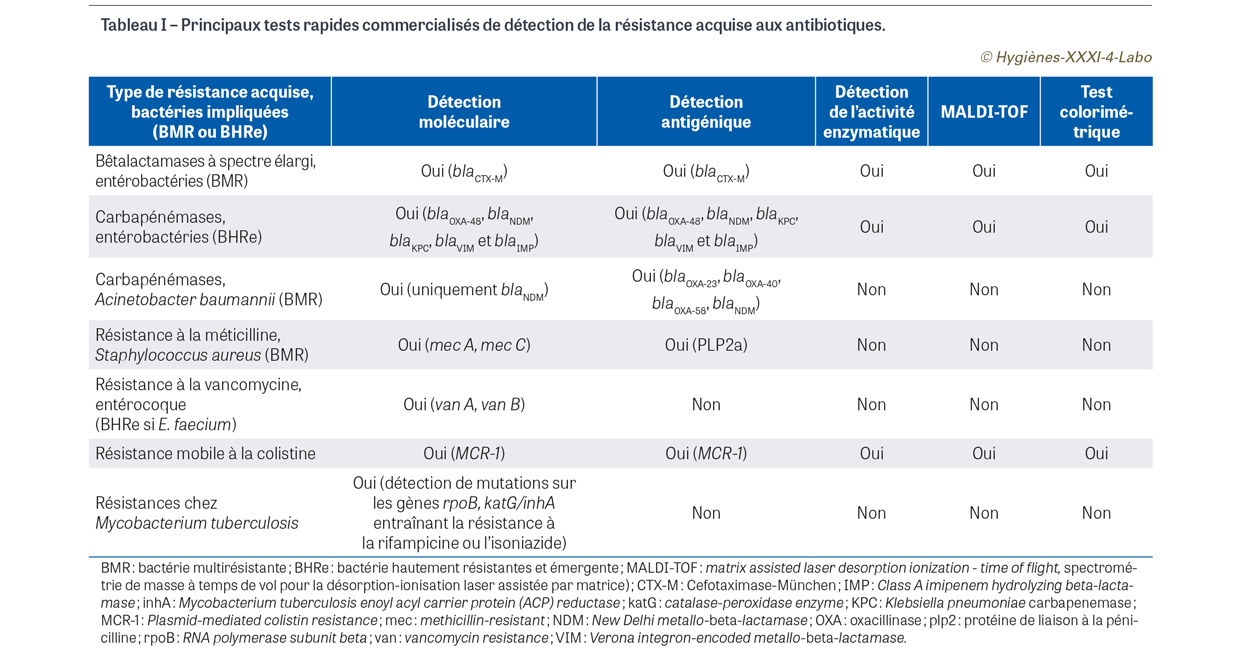
Identification des gènes de résistances
La mise en évidence des gènes codant certaines résistances acquises est une approche rapide (aux alentours d’une heure), notamment lorsqu’elle est automatisée. Ce sont souvent les mêmes trousses qui sont utilisées pour la détection des gènes de résistance directement dans les prélèvements cliniques et que nous avions décrites dans un précédent volet de cette rubrique [4]. La culture obtenue est mise en suspension et traitée comme un échantillon clinique. Les techniques « en temps réel » (real time polymerase chain reaction1 ou RT-PCR) associant une extraction automatisée du matériel génétique et une détection en temps réel des cibles pendant l’amplification sont particulièrement simples à utiliser. Le nombre de cibles recherchées est souvent limité (inférieur ou égal à cinq). D’autres technologies, encore plus sophistiquées, permettent d’élargir le panel de gènes recherchés : elles sont basées, par exemple, sur l’hybridation des produits de PCR ciblés sur des membranes sensibilisées par les séquences complémentaires. Ces deux techniques sont retrouvées pour la détection de la résistance à la vancomycine chez les entérocoques, de la résistance à la méticilline chez Staphyloccoccus aureus, ou encore de la résistance à la rifampicine chez Mycobacterium complex tuberculosis. Dans ce dernier cas, M. tuberculosis étant une bactérie à croissance lente, l’identification d’une résistance à la rifampicine par test rapide à partir d’une culture en milieu liquide est d’autant plus bénéfique pour la prise en charge du patient qu’elle permet l’adaptation thérapeutique immédiate dans l’attente du résultat complet de l’antibiogramme classique, lequel nécessite de quatre à huit semaines. La résistance à la rifampicine est en effet le plus souvent associée à la résistance à l’isoniazide : les souches de M. tuberculosis sont alors considérées comme multirésistantes aux antibiotiques et nécessitent des traitements et des mesures d’isolement spécifiques (durée…). Concernant les entérobactéries, seule la recherche des principaux gènes de carbapénémases est disponible : blaOXA-48, blaNDM, blaKPC, blaVIM et blaIMP2. En France, les trois principales carbapénémases détectées étant blaOXA-48, blaNDM et blaVIM, les cibles choisies par les industriels sont donc bien adaptées à l’épidémiologie nationale [9]. Néanmoins, en cas de mutation sur l’un des sites de fixation des amorces nécessaires à l’amplification des gènes, la réaction devient faussement négative. Outre le risque de ne pas identifier un gène peu commun (il existe plusieurs milliers de bêtalactamases décrites), il ne faut pas oublier que la présence d’un gène n’implique pas forcément son expression, donc la présence de la résistance phénotypique chez la bactérie concernée. De plus, chez certaines espèces bactériennes, la résistance est liée à la surexpression d’un gène naturellement présent (par exemple certaines souches d’A. baumannii résistantes à l’imipénème [ABRI] par surexpression de l’oxacillinase naturelle blaOXA-51) : sa simple présence ne suffit pas à identifier un phénotype de résistance acquise. Ces tests coûtent quelques dizaines d’euros et nécessitent souvent un appareillage spécifique qui peut néanmoins être utilisé pour d’autres tests de diagnostic moléculaire.
Identification des protéines impliquées dans la résistance
La mise en évidence de la protéine responsable de l’acquisition d’une résistance est une approche qui s’est très largement démocratisée ces dix dernières années. Les cibles recherchées étant principalement des enzymes de nature glycoprotéique, il est facile d’obtenir des anticorps monoclonaux spécifiques capables de se lier à ces structures ; les liaisons anticorps-antigènes sont ensuite révélées par des techniques très bien maîtrisées en immunologie (Figure 2). De plus, ces techniques ont évolué : après l’agglutination de particules de latex parfois difficile à interpréter, les approches dites immunochromatographiques proposent une migration de l’échantillon sur membrane avec détection de traits de lecture bien lisibles ; elles permettent en outre la détection de plusieurs cibles en même temps. Ces réactifs sont disponibles pour rechercher la résistance à la méticilline chez S. aureus (mise en évidence de la protéine PLP2a3), des carbapénémases chez les entérobactéries (liste identique à celles ciblées par les approches moléculaires) [10], des carbapénémases chez les ABRI (OXA-23, OXA-40, OXA-51, NDM) [11], la principale classe de bêtalactamases à spectre étendu (BLSE) chez les entérobactéries (céfotaximases de type CTX-M4), et le variant MCR-15 de l’enzyme impliquée dans la résistance plasmidique d’Escherichia coli à la colistine [12] (Tableau I).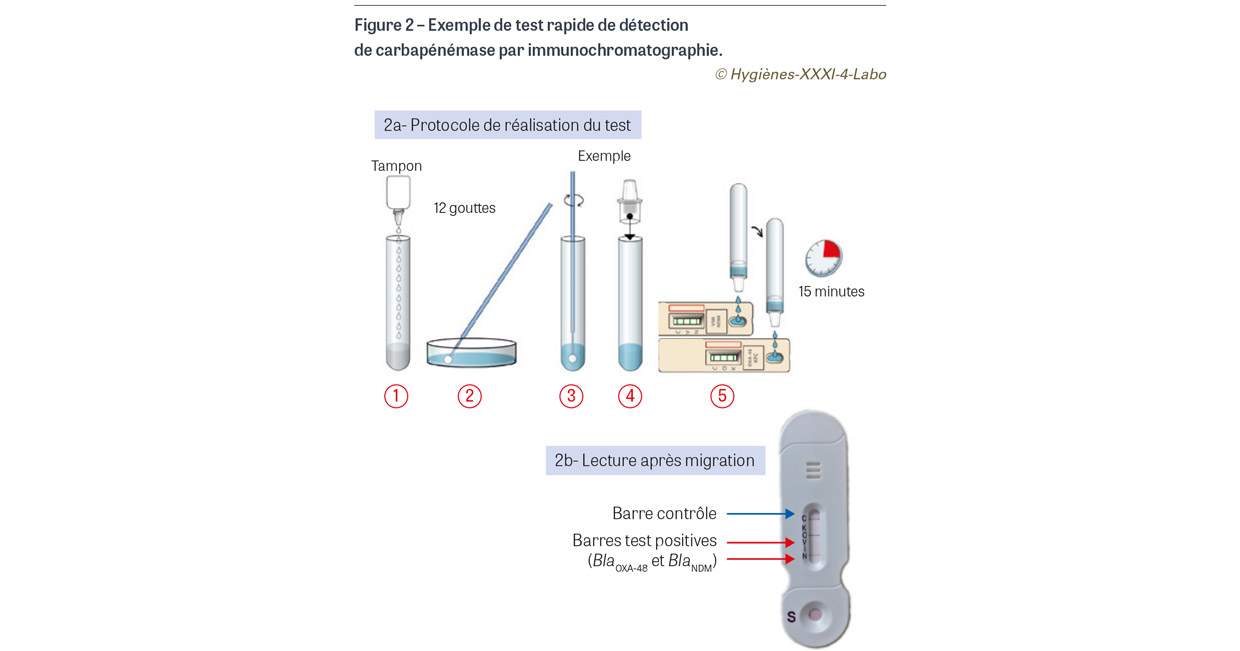
Détection enzymatique de résistances acquises par hydrolyse du substrat
La majorité des mécanismes de résistance aux bêtalactamines acquis par les bactéries est liée à la présence d’enzymes hydrolysant (et ainsi « inactivant ») les bêtalactamines (pénicillines, céphalosporines et carbapénèmes) : parmi celles-ci sont retrouvées les BLSE et les carbapénémases. Ces tests rapides sont donc essentiellement utilisés pour détecter les résistances à ces molécules chez les bactéries à coloration de Gram négative telles que les entérobactéries, P. aeruginosa ou encore A. baumannii. Leur intérêt principal est qu’ils détectent l’ensemble des enzymes exprimant l’activité catalytique recherchée, quel que soit leur type : ces tests sont donc plus sensibles que ceux ne détectant que quelques types précis de cibles comme les tests moléculaires et immunologiques. Dans un second temps, l’identification précise de l’enzyme impliquée est importante à la fois pour optimiser le traitement antibiotique en attendant l’antibiogramme complet et pour détecter un début d’épidémie liée au même support génétique.
Tests chromogéniques
La présence de ce type d’enzyme chez une bactérie peut être détectée en mettant son contenu en contact avec un substrat chromogène, c’est-à-dire qui change de couleur s’il est hydrolysé. Depuis très longtemps de tels tests permettent de rechercher la présence de pénicillinase chez S. aureus : ils sont basés sur la nitrocéfine, qui passe d’une couleur jaune à une couleur rouge lorsqu’elle est hydrolysée [13]. Des substrats chromogéniques ont été développés pour rechercher les BLSE et les carbapénémases [14,15]. La réalisation de ces deux derniers tests est rapide et très simple : l’étape d’extraction de l’enzyme et la révélation de son activité nécessitent la mise en suspension de quelques colonies dans un mélange d’une ou plusieurs gouttes de deux réactifs. La lecture finale est visuelle en une dizaine de minutes : le substrat change de couleur en cas de résultat positif (Figure 3). Les performances de ces réactifs sont excellentes. Néanmoins d’authentiques enzymes très peu actives peuvent donner lieu à des tests faussement positifs. À l’inverse, des enzymes non recherchées mais produites en très grande quantité (par exemple les céphalosporinases hyperproduites) peuvent être à l’origine de réactions positives ; il est donc intéressant de bien caractériser en parallèle l’espèce de la souche testée afin d’interpréter un résultat faiblement positif au regard de la présence naturelle d’une céphalosporinase comme chez Enterobacter cloacae, Klebsiella aerogenes, Citrobacter freundii ou Morganella morganii. Concernant la détection des carbapénémases, le principal test chromogénique disponible ne détecte pas certaines enzymes assez rarement retrouvées en France (blaIMI, blaNMC-A, blaSME et blaFRI6).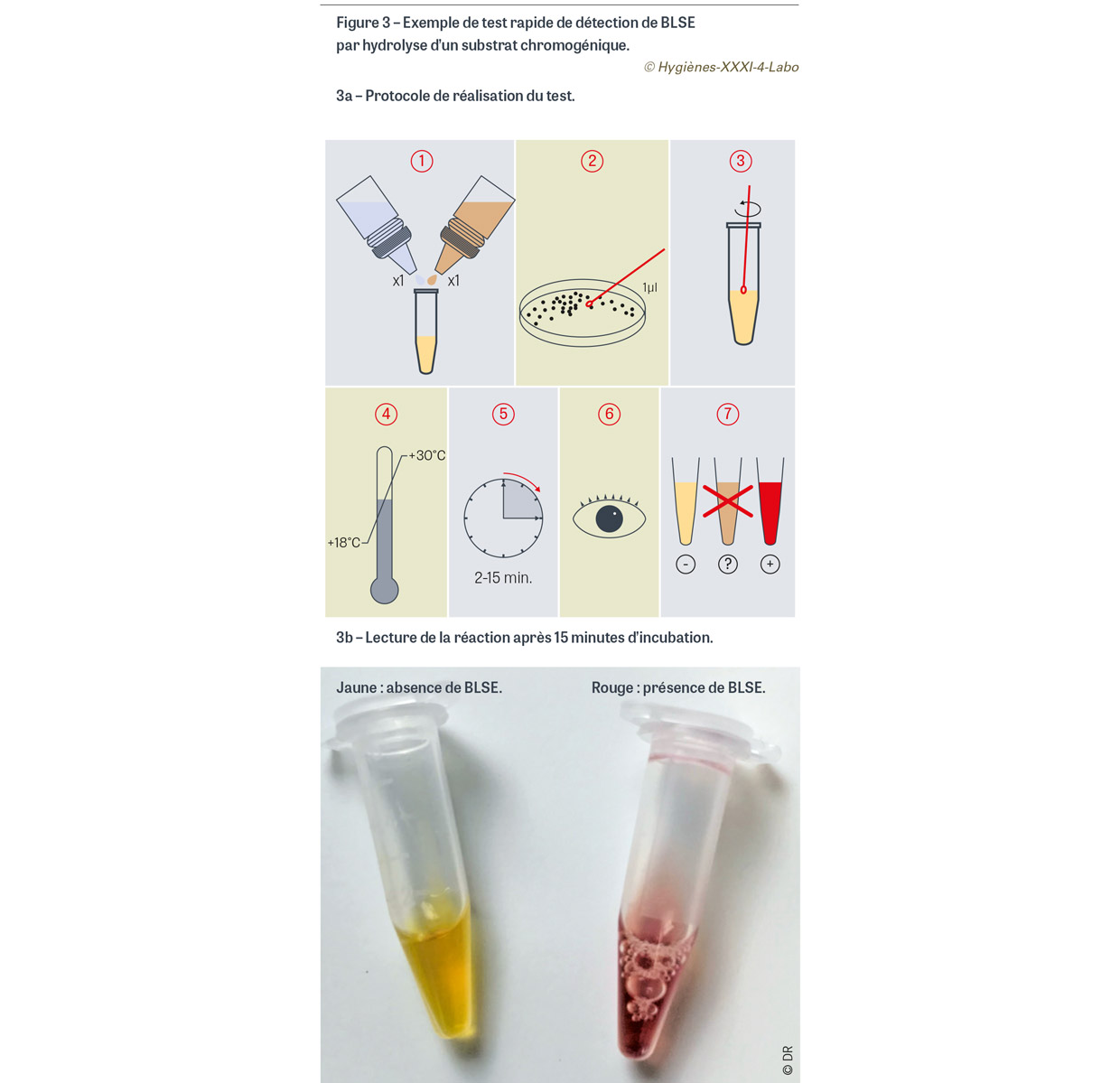
Tests d’hydrolyse simple
Les tests d’hydrolyse simples sont basés sur le même principe que précédemment mais ne font pas appel à des substrats chromogènes. Le substrat est constitué de la molécule de référence (céfotaxime, imipénème), ce qui peut être considéré comme un avantage analytique.
Tests unitaires
L’hydrolyse est révélée par l’acidification du milieu à l’aide d’un indicateur coloré (rouge de phénol) qui change de couleur en milieu acide (Figure 4).
Tests utilisant la technologie MALDI-TOF
La technologie MALDI-TOF (matrix assisted laser desorption ionization - time of flight7) est basée sur le principe de la spectrométrie de masse associée qui permet d’identifier des structures chimiques après leur bombardement par des électrons et la séparation des produits de dégradation par l’analyse de leur taille. La technologie MALDI-TOF utilise un rayon laser UV pour générer des produits de dégradation qui seront ensuite analysés. Cette technologie disponible depuis plus de vingt ans s’est démocratisée et équipe désormais la grande majorité des laboratoires de microbiologie qui l’utilisent pour identifier les bactéries, les levures et les champignons filamenteux. Dans le cadre des résistances bactériennes, la technologie MALDI-TOF peut être utilisée pour la détection de carbapénémases. La technologie MALDI-TOF analyse la molécule testée avant et après sa mise en contact pendant quelques minutes avec l’extrait bactérien : en présence de l’enzyme recherchée, le pic correspondant à la masse du substrat disparaît au profit de l’apparition de pics correspondants aux produits de dégradation, ce qui va générer un changement de spectre [16].
Détection de la résistance phénotypique par culture en milieu additionné d’antibiotique
La souche bactérienne à tester est mise en culture en présence et en l’absence d’une concentration critique d’antibiotique (concentration à partir de laquelle la souche est considérée comme résistante) : la capacité de multiplication de la souche en présence de l’antibiotique (donc sa résistance) est révélée par l’utilisation d’un sucre qui entraîne l’acidification du milieu, ou d’un indicateur de la viabilité des bactéries comme la résazurine. Ces tests sont commercialisés pour la détection de la résistance à la colistine chez les entérobactéries, P. aeruginosa et A. baumannii [11,17].
Conclusion
L’identification rapide des bactéries et des levures en culture étant désormais obtenue en moins de quinze minutes grâce à la technique MALDI-TOF, les industriels et les microbiologistes se focalisent désormais sur l’accélération de l’obtention de résultats de sensibilité aux antimicrobiens. De nombreuses solutions existent déjà, de coût et d’accessibilité variables mais offrant de très bonnes performances analytiques. Les études, très nombreuses, ont montré l’impact de ces techniques sur la rapidité d’obtention des résultats et le délai de la mise en place d’une antibiothérapie efficace. Elles peinent néanmoins à montrer une efficacité dans la diminution de la mortalité qui est un paramètre multifactoriel, notamment très lié au terrain du patient [8]. Concernant la prévention des épidémies, les études manquent pour montrer l’impact direct de ces tests, là aussi du fait du caractère habituellement multifactoriel de la survenue des transmissions croisées : délai d’obtention des résultats bien évidemment, mais aussi disponibilité de chambres individuelles et moyens humains pour mettre en place efficacement les précautions complémentaires. Les délais d’obtention des résultats de ces tests rapides doivent également tenir compte de la présence de personnel de laboratoire capable de valider et communiquer les résultats en temps utile, c’est-à-dire quand le clinicien ou l’hygiéniste est présent pour mettre en œuvre les mesures adaptées. Dans ce contexte, les tests unitaires rapides dont les résultats sont disponibles en moins d’une heure sont à favoriser. Un dialogue constructif entre microbiologistes, cliniciens et hygiénistes est souhaitable pour identifier les tests et les organisations les plus performantes afin d’accélérer un processus décisionnel qui modifiera le fonctionnement des services et améliorera l’efficacité des mesures de prévention des épidémies. Néanmoins, la vigilance de tous les acteurs reste de mise en raison de la formidable capacité d’adaptation des bactéries aux antibiotiques par la sélection de nouveaux mécanismes de résistance qui ne seront donc potentiellement pas détectés par certains tests rapides.
Notes :
1- Réaction de polymérisation en chaîne en temps réel.
2- OXA : oxacillinase ; NDM : New Delhi metallo-beta-lactamase ; KPC : Klebsiella pneumoniae carbapenemase ; VIM : Verona integron-encoded metallo-beta-lactamase ; IMP : Class B imipenem hydrolyzing beta-lactamase.
3- Protéine de liaison à la pénicilline additionnelle.
4- Cefotaximase-München.
5- Plasmid-mediated colistin resistance, résistance à la colistine médiée par plasmide.
6- IMI : Class A imipenem hydrolyzing beta-lactamase ; NMC-A : not metalloenzyme carbapenemase class A ; SME : Serratia marcescens enzyme ; FRI : Fribourg carbapenemase.
7- [Spectrométrie de masse] à temps de vol pour la désorption-ionisation laser assistée par matrice.