Pour ce premier volet, nous allons aborder les tests PCR (ou Polymerase chain reaction1) réalisables directement sur un prélèvement clinique et ciblant différents pathogènes ou marqueurs en même temps. Ils sont regroupés sous le nom de PCR multiplexes : différentes cibles génétiques sont recherchées, amplifiées et détectées simultanément. Les micro-organismes recherchés en même temps peuvent être des bactéries, des virus, ou encore des champignons ou des parasites responsables d’un même syndrome (c’est-à-dire d’un ensemble de symptômes). Ces tests proposent des panels syndromiques d’amorces ciblant des agents infectieux (parfois associés à des gènes de résistance aux antimicrobiens ou à des gènes de virulence) à l’origine d’une même pathologie [1]. Nous nous intéresserons dans cet article aux tests entièrement automatisés qui ont contribué à démocratiser cette technique dans de nombreux laboratoires. L’épidémie de Covid-192 a significativement renforcé la diffusion de ces tests dans les laboratoires de biologie, qui se sont largement équipés d’appareils permettant ce genre d’approche. Nous exclurons de cet article les tests PCR ne ciblant qu’un seul pathogène, même si plusieurs cibles sont incluses dans le réactif (par exemple : recherche des toxines de Clostridium difficile, recherche de Staphylococcus aureus et du gène de résistance à la méticilline). La quasi-totalité des réactifs de PCR commercialisés contiennent d’ailleurs un contrôle interne d’amplification qui fait d’eux des PCR multiplexes par définition. Nous nous intéresserons néanmoins aux PCR multiplexes de détection des bactéries hautement résistantes et émergentes (BHRe), domaine d’intérêt particulier pour les hygiénistes.
Description de l’offre disponible
Les PCR multiplexes, c’est-à-dire recherchant plusieurs cibles en même temps dans un même prélèvement avec un seul test, existent depuis plusieurs dizaines d’années. C’est leur automatisation et leur simplification à l’extrême qui ont constitué un tournant pour le microbiologiste, le clinicien et l’hygiéniste. De plus, les approches syndromiques ont transgressé les séparations entre les différentes spécialités de la microbiologie et les laboratoires correspondants. Les analyses sont réalisées sur des plateformes de plus en plus compactes réalisant de façon automatisée les trois étapes des tests PCR (ou apparentés) : extraction, amplification et révélation de l’ADN3 ou de l’ARN4 des micro-organismes recherchés. Ces tests peuvent être réalisés par un personnel formé a minima n’ayant aucune spécialisation en biologie moléculaire, ce qui permet leur réalisation sur des plateaux techniques polyvalents fonctionnant 24 heures sur 24 et 7 jours sur 7, ou dans des structures plus petites de type « point of care5 » éloignées géographiquement des plateaux techniques. Leur délocalisation à l’intérieur même des services cliniques et leur utilisation par du personnel soignant n’ayant pas de formation de laboratoire sont techniquement possibles. En effet, en plus de leur miniaturisation et de leur automatisation, ces tests sont associés à des logiciels qui donnent des résultats validés techniquement et interprétés (présence ou absence de la cible), directement utilisables par les cliniciens. La biologie délocalisée, initialement limitée à des tests simples (biochimie d’urgence, tests immunologiques basiques, voire test génomique ciblant un seul pathogène), prend ainsi une nouvelle dimension. Les panels syndromiques concernent par exemple les infections neuro-méningées, les infections respiratoires hautes ou basses, les infections gastro-
intestinales, les infections sexuellement transmissibles, les infections digestives et intra-abdominales, les infections ostéo-articulaires… (Tableau I). Des panels sont également commercialisés pour être utilisés sur des flacons d’hémoculture identifiés comme positifs par un système automatisé : ils ciblent des bactéries, des levures et des gènes de résistance aux antibiotiques. Comme évoqué précédemment, nous incluons à cette liste les PCR multiplexes recherchant plusieurs gènes de résistance dans un prélèvement de selles, utiles pour le dépistage des porteurs de BHRe. Les résultats sont obtenus très rapidement, en une heure environ.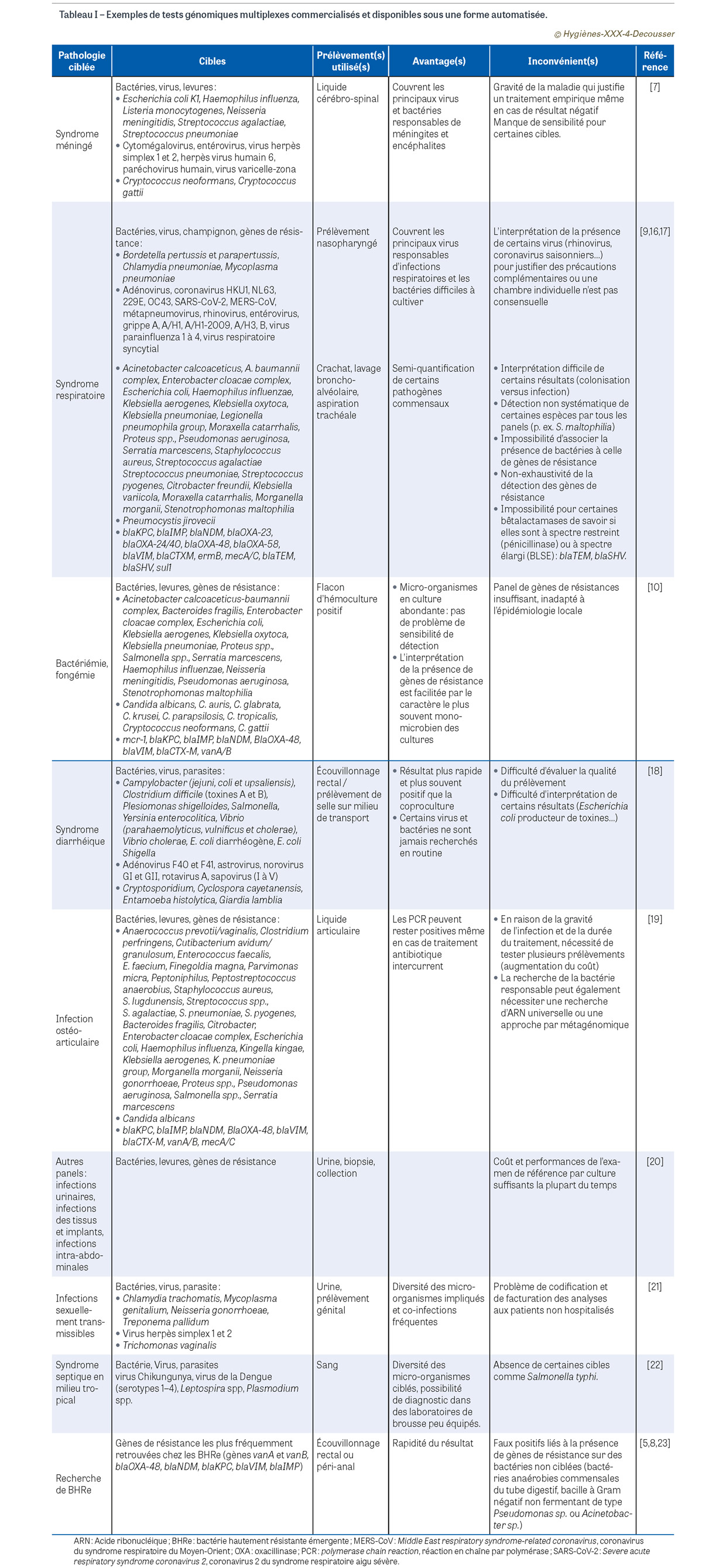
Performances analytiques et limites de l’approche
Les performances analytiques, c’est-à-dire la capacité à détecter la présence de la cible (sensibilité) et uniquement cette cible (spécificité), varient selon les trousses commerciales et, à l’intérieur d’un panel, en fonction du micro-organisme recherché. Pour simplifier, un défaut de sensibilité entraîne des résultats faussement négatifs et une mauvaise valeur prédictive négative du test (probabilité qu’en effet la cible ne soit pas présente quand le test est rendu « négatif »). Un défaut de spécificité entraîne des résultats faussement positifs et une mauvaise valeur prédictive positive du test (probabilité qu’en effet la cible soit bien présente quand le test est rendu « positif »).
Sensibilité
La sensibilité de ces panels est toujours inférieure à des PCR simples ne recherchant qu’une cible à la fois. Ainsi, pour le panel syndromique « méningites », il a été établi un manque de sensibilité pour la détection des virus herpétiques à l’origine de résultats faussement négatifs [2]. Il en est de même pour la levure Cryptococcus neoformans [3]. Concernant la détection des bactéries multirésistantes, la technique dite « de référence » est la culture ; la sensibilité des tests PCR par rapport aux techniques de culture dépend néanmoins du type de culture réalisé. Il est important de souligner que, dans la quasi-totalité des laboratoires, la sensibilité d’un dépistage par culture réalisé en routine, c’est-à-dire ensemencé directement sur une gélose sélective sans bouillon d’enrichissement, n’est que de 80% environ par rapport à celle obtenue avec une étape supplémentaire d’enrichissement, qui allonge néanmoins le délai de résultat de 24 heures [4]. En fonction de la technique de référence utilisée (culture avec ou sans enrichissement), les tests PCR peuvent avoir une sensibilité égale voire supérieure à la culture (seuil de détection variant de 10 à 1 000 unités formant colonie par millilitre) [5]. Comme pour toutes les techniques de PCR ciblées, « on ne trouve que ce que l’on cherche ». Cela est particulièrement important lorsque le nombre de micro-organismes ou de gènes de résistance est potentiellement très élevé. Ainsi, contrairement aux gènes responsables de la résistance aux béta-lactamines chez S. aureus (mecA et exceptionnellement mecC) et à la vancomycine chez les entérocoques (vanA et vanB), les gènes codant les bêtalactamases sont extrêmement nombreux (plus de 7 0006). Les industriels doivent donc faire des choix dont la pertinence varie en fonction de l’adéquation à l’épidémiologie locale : ainsi la présence de la carbapénémase KPC7 que l’on retrouve dans de nombreux kits est due à sa forte prévalence en Amérique du Nord alors qu’elle est très peu fréquente en France. De même, la formulation initiale d’une des principales trousses de détection des carbapénémases ne comprenait pas OXA-488 car cette enzyme est très peu fréquente en Amérique du Nord [5]. De plus, quelques mutations situées sur les sites de fixation (hybridation) des amorces permettant l’amplification des gènes cibles suffisent à rendre le test faussement négatif. Ce fut le cas pour le variant OXA-181 des carbapénémases de la famille des OXA-48, non détectées par les amorces initialement choisies par l’industriel, qui a dû mettre à jour la composition du réactif [5]. La qualité du prélèvement est essentielle aux performances de ces tests. Pour des raisons de simplification des protocoles d’extraction du matériel génétique, les milieux de transport liquides sont privilégiés (par exemple les selles). Cela limite l’évaluation de la qualité du prélèvement de départ : selles diarrhéiques, écouvillon rectal teinté… Concernant les infections pulmonaires, une part importante des prélèvements respiratoires sont réalisés par les cliniciens « à l’aveugle » sans que l’on soit sûr de prélever l’échantillon sur le site anatomique précis de l’infection. Parfois les techniques deviennent trop sensibles : la mise en évidence de la toxine de Clostridium difficile par PCR seule ou au sein d’un panel syndromique a artificiellement fait augmenter l’incidence des diarrhées dues à cette bactérie en détectant de simples porteurs. Cette approche ne doit plus être utilisée en première intention [6].
Spécificité
La spécificité des tests n’est pas non plus parfaite. Par exemple, des faux positifs du panel méningé ont été mis en évidence pour le pneumocoque [7]. La contamination du prélèvement par l’opérateur pouvant être porteur de ces micro-organismes a été évoquée. Concernant la détection de certaines bêtalactamases, il est parfois difficile de différencier, au sein des milliers de types différents, celles d’intérêt comme les carbapénémases des autres à spectre d’activité plus restreint [5]. De plus, la mise en évidence d’un de ces gènes ne signifie pas forcément la présence d’une entérobactérie productrice de carbapénémases : il peut être présent dans une souche de Pseudomonas sp. ou d’Acinetobacter sp. qui ne sont pas à proprement parler des BHRe [5]. Cela est encore plus vrai pour les résultats positifs des recherches du gène vanB dans les selles par PCR qui ne sont associés à une culture positive pour un entérocoque résistant à la vancomycine (ERV) que dans moins de 3% des cas ; pour le gène vanA, un ERV est retrouvé en culture dans les deux tiers des cas lorsqu’une PCR positive a été trouvée [8]. Des gènes vanB non associés à des ERV sont assez fréquents dans des bactéries du microbiote digestif. Hormis dans une situation épidémique à ERV vanB, une PCR positive au gène vanB dans un dépistage rectal n’a donc aucune signification et doit être systématiquement confirmée par une culture. Concernant les prélèvements respiratoires, il faut distinguer les panels respiratoires « hauts » des panels respiratoires « bas ». Les premiers détectent de façon qualitative à partir d’un prélèvement nasopharyngé la présence de bactéries le plus souvent considérées comme des pathogènes stricts (par exemple Bordetella pertussis, bactérie responsable de la coqueluche), mais surtout celle de virus. L’interprétation est simple et purement qualitative (présence ou absence). Les panels de PCR multiplexes « bas » recherchent en plus à partir de prélèvements respiratoires plus profonds des bactéries dont certaines sont potentiellement commensales, c’est-à-dire présentes naturellement (Tableau I). Le résultat rendu est quantitatif à partir d’un nombre de copies de génome par millilitre. Les critères d’interprétation historiques sont basés quant à eux sur la culture quantitative [9]. Même si les deux types de résultats semblent concordants, il faut rester prudent.
Intérêt selon l’acteur
Au-delà des performances de ces tests et de leurs limites analytiques, leur place dans la prise en charge des patients et leur contribution à son amélioration dépend de plusieurs facteurs qui varient selon l’acteur.
Intérêt pour le biologiste
Le biologiste n’a plus besoin de disposer d’un personnel formé à la biologie moléculaire ; il peut proposer des analyses sur des plages horaires très élargies, y compris au sein de laboratoires de garde polyvalents ou de points of care, petites structures délocalisées de la plateforme centrale de biologie, qui peuvent se trouver dans un autre établissement. Il existe néanmoins des inconvénients : les réactifs, souvent très coûteux (de quelques dizaines à centaines d’euros pièce), nécessitent des équipements spécifiques. Pour ne pas devoir les acheter, le biologiste doit s’engager à consommer un nombre de tests prédéfini par l’industriel. Ces réactifs et les machines qui les utilisent peuvent être source de dépendance et de défaillance en cas de rupture d’approvisionnement. Si le biologiste veut pouvoir disposer de plusieurs techniques pour la même recherche, il doit en établir la corrélation auprès de son organisme de certification. Des discordances entre les résultats de deux panels différents peuvent exister, en fonction des spécificités de chacun. Les conséquences de la délocalisation de la biologie au sein des services cliniques évoquées précédemment, ainsi que l’absence de remboursement pour les patients non hospitalisés, peuvent constituer des freins à la généralisation de ces tests. La mise en place d’approches syndromiques associant les différentes spécialités de la microbiologie peut matériellement et parfois politiquement poser des problèmes lorsque ces disciplines existent au sein d’unités séparées géographiquement ou budgétairement. Enfin, certains biologistes regrettent une détérioration du raisonnement clinique ciblant une étiologie précise au profit d’une approche large de criblage, moins réfléchie.
Intérêt pour le clinicien
Le clinicien peut être demandeur de ces tests rapides afin d’être conforté dans son choix thérapeutique à la fois efficace et respectueux du microbiote du patient et de l’écologie microbienne de son service. La mise en évidence de virus permet désormais également la prescription de traitements antiviraux ciblés (grippe, Covid-19). Néanmoins, le délai lié à l’acheminement du prélèvement peut être supérieur à la phase analytique, qui dure souvent moins d’une heure. Il doit également être sensibilisé par le biologiste à la présence de « trous » dans les panels proposés : par exemple, en cas de pneumopathie tardive acquise sous ventilation, l’espèce Stenotrophomonas maltophilia, pourtant impliquée notamment chez les patients multitraités par antibiotiques, ne fait pas systématiquement partie des cibles incluses dans les panels respiratoires. Il en est de même pour la détection des résistances acquises dont certaines, notamment chez P. aeruginosa, ne sont pas dues à l’acquisition des gènes supplémentaires facilement détectables mais à des surexpressions de gènes naturellement présents. L’interprétation de la présence de gènes de résistance à partir d’un prélèvement multimicrobien est quant à elle difficile : le gène de résistance identifié est-il bien associé à la bactérie suspectée d’être responsable de l’infection ? Les panels de PCR multiplexes sont concurrencés par des techniques d’antibiogrammes phénotypiques dites « rapides » qui permettent une identification et un antibiogramme par extrapolation des concentrations minimales inhibitrices en sept heures environ [10]. Ce genre de subtilité nécessite la disponibilité de médecins sensibilisés et aguerris qui sauront traduire ces résultats en une adaptation de la stratégie anti-infectieuse. Dans le cas contraire, notamment en période de garde ou de week-end, il est probable que, si l’état clinique du patient est stable, l’adaptation du traitement antibiotique soit remise au lendemain, ce qui est un comble quand on dispose d’un test rapide. Enfin, la gravité de certaines infections (méningite, choc septique…) incite le clinicien à ne prendre aucun risque et à « couvrir » largement par son choix thérapeutique l’ensemble des micro-organismes potentiellement responsables jusqu’à la preuve définitive de leur absence.
Intérêt pour le patient
En théorie, le patient est le premier bénéficiaire d’un traitement efficace qui respecte aussi son écologie microbienne : un microbiote perturbé constitue un facteur de risque d’acquisition d’une bactérie multirésistante par acquisition exogène ou par sélection endogène [11]. En pratique, les études clinico-biologiques mettant en évidence la diminution du délai de l’obtention des résultats microbiologiques et l’adaptation du traitement sont pléthores, alors que celles évaluant l’intérêt clinique (impact sur la mortalité ou sur la durée de séjour) sont bien moins nombreuses et exceptionnellement favorables. Certaines identifient même parfois un risque d’erreur potentiellement délétère [12]. L’intérêt de la diminution de quelques jours d’antibiotiques sur la sélection de résistance ou l’apparition de surinfection chez le patient n’est pas démontré à ce jour [13]. Une situation particulière est celle de patients éloignés de structures de soins modernes (exemple de certains pays/certaines régions dépourvues de laboratoires équipés) et qui pourraient bénéficier de la réalisation de panel de PCR dans des structures avancées. Cette approche suivrait l’exemple de la détection automatisée de Mycobacterium tuberculosis et de l’identification de mutations associées à la résistance aux antibiotiques qui permet dans des pays ou des zones en voie de développement de s’affranchir d’un laboratoire structuré. Un kit récent permet ainsi sur un prélèvement sanguin d’identifier les virus du chikungunya, de la dengue ainsi que les agents du paludisme et de la leptospirose [22].
Impact pour l’hygiéniste
« Dépister vite pour isoler mieux » pourrait être la devise de ces panels de PCR, permettant à l’hygiéniste d’éviter les épidémies sans multiplier la mise en « précautions complémentaires » qui sont coûteuses en temps et en argent, et moins bien respectées lorsqu’elles se multiplient [14]. La rapidité de la mise en précautions complémentaires des patients porteurs de BHRe a été corrélée à la diminution de la survenue de cas secondaires dans une étude française de qualité [15]. Bien qu’intuitif, l’impact direct des techniques rapides de détection des porteurs de bactéries multirésistantes aux antibiotiques (BMR) ou de BHRe sur la survenue d’épidémie n’est toujours pas strictement établi à ce jour. Concernant la prise en charge de certains syndromes infectieux, l’identification d’un pathogène, même sans conséquence thérapeutique, pourrait entraîner un meilleur respect des précautions complémentaires par rapport à un syndrome non microbiologiquement étayé. Cependant, la mise à disposition de panels respiratoires larges peut être à l’origine de certaines questions : si le panel est négatif mais que le patient présente un syndrome respiratoire potentiellement infectieux, le maintien des précautions complémentaires, pourtant justifié, est parfois plus difficile à faire accepter aux cliniciens. Par ailleurs, la conduite à tenir devant l’identification d’un micro-organisme « banal » comme le rhinovirus ne fait pas consensus. Plus généralement, l’intérêt et la rentabilité de ces tests rapides, indépendamment de leurs performances analytiques et de la rapidité de l’obtention des résultats, sont étroitement liés à la disponibilité des moyens d’action : chambres individuelles, personnel en nombre suffisant pour respecter les mesures adaptées (précautions complémentaires, marche en avant, sectorisation). Dans le cas contraire, la contribution de ces tests sera, in fine, limitée.
Conclusion
Les tests microbiologiques rapides, et plus particulièrement les PCR multiplexes, constituent un outil particulièrement intéressant pour la promotion du bon usage des antibiotiques et la prévention de la transmission croisée. L’hygiéniste doit en connaître les avantages mais également les limites pour les utiliser ou les interpréter à bon escient. Il faut par ailleurs que leur impact sur la qualité de la prise en charge soit mieux établi scientifiquement afin d’en justifier le coût. De plus ils doivent s’accompagner d’une organisation humaine et matérielle suffisante pour permettre leur plein rendement.
Notes :
1- Réaction en chaîne par polymérase.
2- Coronavirus disease 2019, maladie à coronavirus 2019.
3- Acide désoxyribonucléique.
4- Acide ribonucléique.
5- Point of care test : test sur le lieu de soin.
6- Voir : http://www.bldb.eu
7- Klebsiella pneumoniae carbapenemase.
8- Oxacillinase 48.