Introduction
L’antibiogramme est un des tests du laboratoire de microbiologie les plus attendus par les cliniciens : c’est le test qui leur permet de prescrire de façon éclairée une antibiothérapie ciblée et théoriquement efficace sur la bactérie responsable de l’infection. Il est souvent attendu comme le « Graal », bien plus que les autres résultats d’analyses microbiologiques.
L’antibiogramme : pourquoi ?
Il existe deux types de résistance à un antibiotique chez les bactéries. Dans le cas d’une résistance naturelle, toutes les souches d’une même espèce bactérienne sont résistantes à cet antibiotique. La raison est souvent une caractéristique constitutionnelle de l’espèce qui empêche l’antibiotique d’agir (paroi ou membrane externe de la bactérie empêchant l’antibiotique d’entrer dans la bactérie, présence naturelle d’une enzyme qui détruit l’antibiotique ou d’une pompe d’efflux qui l’éjecte dès qu’il entre à l’intérieur de la bactérie). Il suffit donc d’identifier l’espèce bactérienne pour savoir quels antibiotiques sont à exclure du choix thérapeutique (Tableau I). Dans le cas d’une résistance acquise, seule une partie des souches au sein de l’espèce bactérienne, a acquis ou développé une résistance à un antibiotique. Les mécanismes impliqués sont multiples : modification de la cible des antibiotiques (résistance du pneumocoque et de Staphylococcus aureus aux pénicillines et aux céphalosporines) ; destruction de l’antibiotique par une enzyme (résistance des entérobactéries aux céphalosporines de troisième génération et aux carbapénèmes) ; diminution ou suppression de la pénétration de l’antibiotique dans la bactérie qui ne peut alors plus atteindre sa cible (résistance de Pseudomonas aeruginosa aux carbapénèmes) ; expulsion de l’antibiotique entré dans la bactérie vers l’extérieur par des pompes d’efflux (résistance de Pseudomonas aeruginosa à de nombreuses classes d’antibiotiques) ; protection de la cible des antibiotiques (résistance de S. aureus à l’acide fusidique).
Par ailleurs, ces résistances reposent sur plusieurs types de mécanismes ou supports génétiques. Les mutations surviennent spontanément lors de la réplication du matériel génétique des bactéries. Pour mémoire, une bactérie comme Escherichia coli se réplique toutes les vingt minutes. Parmi les erreurs produites lors de la duplication de l’ADN1, certaines sont corrigées par la bactérie, d’autres conduisent à la mort de celle-ci, et d’autres encore lui donnent par hasard un avantage sélectif comme le développement d’une résistance à un antibiotique. La résistance aux antibiotiques acquise par mutation est ensuite transmise verticalement aux générations descendantes de la souche mutée. Autre possibilité, l’acquisition de matériel génétique mobile : la quasi-totalité des antibiotiques utilisés en médecine humaine proviennent de molécules naturelles fabriquées par d’autres bactéries ou par des champignons. Des gènes de résistance préexistent dans ces bactéries environnementales sans intérêt médical. Lors de l’utilisation à large échelle d’antibiotiques dérivés de ces composés naturels, leurs gènes de résistance peuvent être mobilisés vers des bactéries d’intérêt médical à partir de différents supports génétiques mobiles comme des plasmides, des transposons ou des intégrons. Ces supports génétiques mobiles peuvent être à l’origine de la diffusion rapide et étendue de gènes de résistance. Ils peuvent ainsi être transmis de manière horizontale entre souches bactériennes d’une même espèce ou d’espèces différentes, et ils sont également transmis verticalement aux générations descendantes de la souche ayant acquis le support génétique de la résistance acquise. Ces supports génétiques mobiles de gènes de résistance acquise aux antibiotiques sont particulièrement à redouter en contexte hospitalier du fait de leur capacité à diffuser largement au sein des espèces bactériennes impliquées dans les infections et épidémies associées aux soins.
Pour identifier une population bactérienne ayant développé une ou plusieurs résistances acquises, un antibiogramme est réalisé : il permet de tester l’efficacité des différentes molécules et classes d’antibiotiques auxquelles l’espèce bactérienne en question est habituellement sensible. La présence d’une souche résistante n’est pas prévisible, même si la consommation de l’antibiotique en question dans les mois précédents ou l’inefficacité clinique d’un traitement en cours constituent des facteurs de risque d’identification d’une souche résistante (Tableau I). Pour une espèce bactérienne donnée, la fréquence des résistances acquises varie avec le temps et le lieu (pays, région, hôpital, service de soins…) mais aussi avec le mode d’hébergement du patient : domicile, hôpital de court, moyen ou long séjour, établissement d’hébergement pour personnes âgées dépendantes (Ehpad). Ainsi, à titre d’exemple, la proportion de souches de S. aureus résistant à la méticilline (SARM) était en moyenne de 12,3% parmi toutes les souches de S. aureus isolées lors de prélèvements diagnostiques en France en 2021, mais elle atteignait 44,8% parmi celles issues de patients hébergés en unité de soins de longue durée [1]. L’antibiogramme est donc un test microbiologique qui permet d’identifier, pour une souche bactérienne donnée, les résistances acquises aux antibiotiques. Le choix des antibiotiques testés est donc spécifique à l’espèce bactérienne à laquelle appartient la souche pour laquelle le profil de sensibilité aux antibiotiques est à déterminer. Pour chaque espèce d’intérêt médical, la liste des antibiotiques à tester est précisée dans les référentiels du comité de l’antibiogramme de la Société française de microbiologie [2].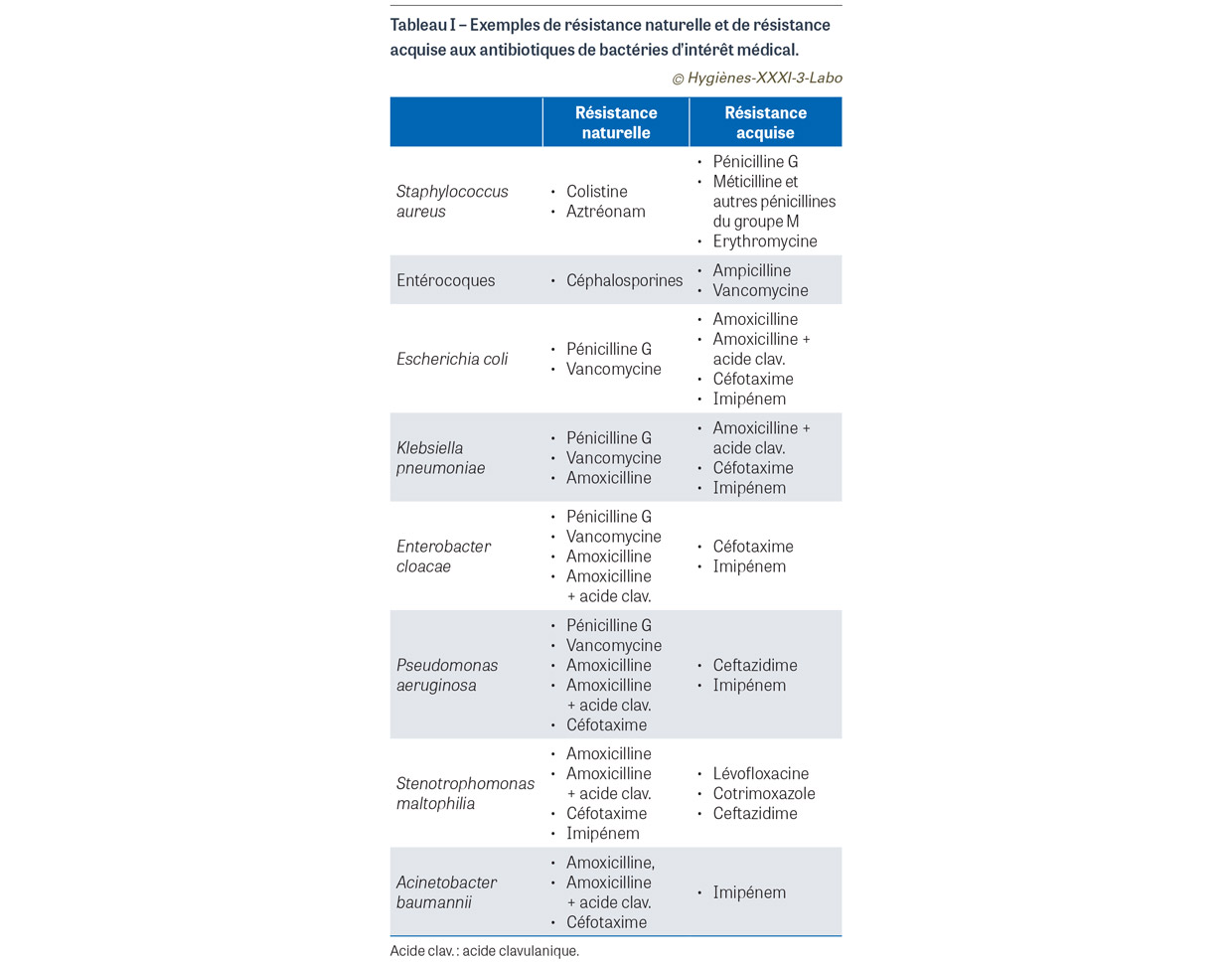
Place de l’antibiogramme dans la prise en charge thérapeutique d’une infection
Comme rapporté précédemment, l’antibiogramme joue un rôle stratégique dans le choix d’une antibiothérapie adaptée. La définition de cette dernière associe la notion d’efficacité sur la souche bactérienne incriminée dans l’infection mais aussi, ce qui est plus récent, le respect des microbiotes du patient, en ayant un impact limité sur les autres bactéries naturellement présentes chez ce dernier. En effet, quoiqu’efficace à court terme, la prescription d’une antibiothérapie à large spectre, c’est-à-dire active sur de nombreux types différents de bactéries, aurait des conséquences délétères à la fois individuelles et collectives. Conséquences individuelles d’une antibiothérapie trop large car la pression de sélection des antibiotiques sélectionne des bactéries résistantes qui, lors du prochain épisode infectieux, pourraient remplacer les bactéries sensibles. De plus, la détérioration des microbiotes présents chez le patient, et notamment son microbiote digestif composé de très nombreuses bactéries anaérobies, favorise le développement d’infections à Clostridioides difficile et la colonisation par des bactéries multirésistantes présentes dans l’environnement du patient. Conséquences collectives d’une antibiothérapie trop large parce que la pression antibiotique favorise la diffusion dans un service hospitalier des bactéries multirésistantes qui prennent la place laissée libre par les bactéries sensibles détruites par les antibiotiques.
Lorsqu’un patient présente une suspicion d’infection, le clinicien devrait idéalement pouvoir disposer des résultats des analyses des prélèvements à visée diagnostique microbiologique avant de prescrire des antibiotiques (hémoculture, examen cytobactériologique des urines [ECBU] ou des crachats [ECBC]…). Cependant, comme nous le verrons plus loin, l’obtention de ces résultats requiert un délai de 48 heures avec les méthodes ordinaires. Ce délai n’est pas compatible avec la prise en charge du patient, et peut même hypothéquer ses chances de survie en cas d’infection grave comme le choc septique pour lequel l’urgence thérapeutique se chiffre en fractions d’heures. Pour ces raisons, le clinicien prescrit le plus souvent une antibiothérapie de première ligne non basée sur les résultats bactériologiques du patient, en tenant compte du contexte clinique du patient (âge, présence de comorbidité comme un diabète ou une immunodépression, grossesse en cours…), de la gravité de l’infection et donc de l’urgence à traiter, des antécédents de prise d’antibiotique dans les mois précédents, de la connaissance d’antécédents d’infection ou de portage de bactéries multirésistantes, du site anatomique impliqué dans l’infection (arbre urinaire, respiratoire…), du type de bactéries habituellement en cause dans l’infection suspectée, de la fréquence habituelle de leurs résistances acquises, et éventuellement des premiers résultats microbiologiques disponibles le jour même, comme l’examen direct d’un ECBU ou d’une hémoculture rapidement positive.
Dans l’attente des résultats complets des analyses microbiologiques, l’antibiothérapie de première intention ainsi prescrite est souvent à large spectre afin de couvrir toutes les bactéries potentiellement impliquées dans l’infection suspectée. Cette antibiothérapie de première intention est appelée antibiothérapie empirique (basée sur l’expérience que l’on a de ce type d’infection) ou probabiliste car basée sur la probabilité d’identifier un type de bactéries avec une résistance acquise : on parle alors de « pari microbiologique » (Figure 1). La valeur acceptable de cette probabilité d’être efficace est de 90%. Pour aider le clinicien dans son choix, des protocoles d’antibiothérapie empirique sont rédigés et diffusés dans chaque hôpital par le comité des agents anti-infectieux. Par exemple, une femme enceinte se présentant avec de la fièvre et une suspicion d’infection urinaire haute recevra du céfotaxime (céphalosporine de troisième génération) par voie intraveineuse, car la principale bactérie impliquée dans ce contexte est Escherichia coli qui est sensible dans plus de 90% des cas au céfotaxime, avec une très bonne diffusion de cette molécule dans les urines. Une fois cette antibiothérapie empirique débutée, il est très important de la réévaluer et de l’adapter aux résultats complets des analyses des prélèvements microbiologiques prescrits et réalisés idéalement avant de débuter l’administration des antibiotiques. Cette réévaluation à 48 heures, délai qui correspond à l’obtention de l’antibiogramme, est appelée « désescalade antibiotique ». Elle doit être tracée dans le dossier du patient et fait l’objet d’indicateurs de performance évalués par exemple par la Haute Autorité de santé [3]. Dans l’exemple proposé précédemment, l’ECBU a permis l’isolement d’E. coli dont l’antibiogramme a montré une sensibilité au céfotaxime mais également à l’amoxicilline, qui est une autre bêtalactamine ayant un spectre d’action plus étroit que le céfotaxime (qui épargne donc mieux le microbiote de la patiente) et qui par ailleurs peut être administrée par voie orale, ce qui limite les risques liés à l’utilisation des dispositifs invasifs. La réévaluation à 48 heures a donc permis de remplacer le céfotaxime par l’amoxicilline pour la suite du traitement antibiotique.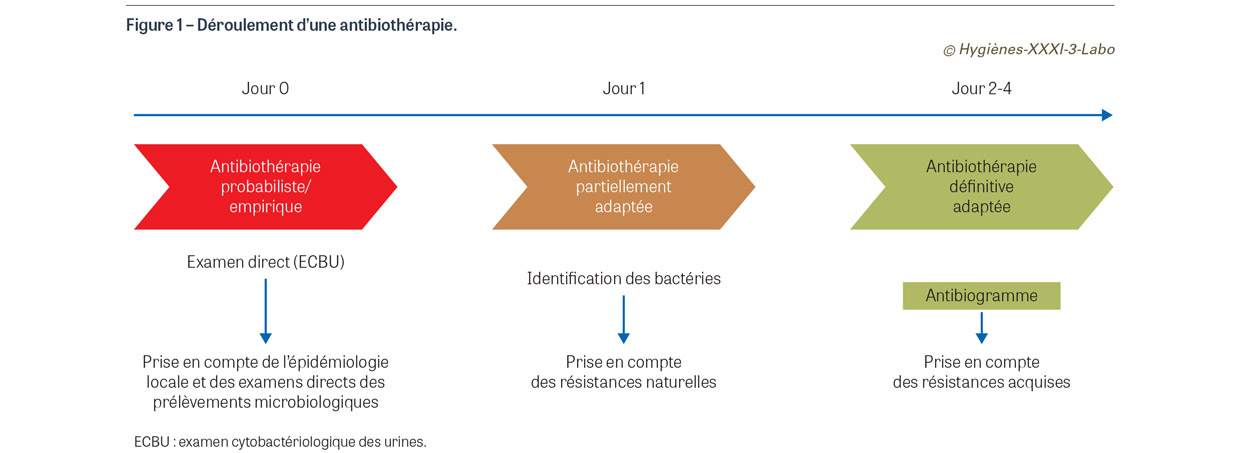
L’antibiogramme : comment ?
L’antibiogramme est un test microbiologique évaluant les éventuelles résistances acquises d’une souche bactérienne aux antibiotiques auxquels elle est naturellement sensible en l’absence de résistance acquise et qui sont donc théoriquement utilisables pour la traiter. Pour que les résultats ainsi obtenus soient robustes, corrélés à la réalité clinique et reproductibles d’un laboratoire à un autre, il existe des instances dans chaque pays qui émettent des recommandations concernant leur réalisation technique et leur interprétation. En France, un comité spécifique de la Société française de microbiologie, le comité de l’antibiogramme (CA-SFM mentionné plus haut), travaille en collaboration avec une commission européenne, l’European Committee of Antimicrobial Susceptibility Testing (EUCAST), qui vise à harmoniser les règles en matière d’antibiogramme entre les différents pays européens. Une commission équivalente existe aux États-Unis, le Clinical and Laboratory Standards Institute (CLSI) [3,4,5].
Le paramètre de base pour évaluer la sensibilité (ou au contraire la résistance) d’une bactérie à un antibiotique est la concentration minimale inhibitrice (CMI) de cet antibiotique vis-à-vis de la bactérie testée : c’est la plus petite concentration d’antibiotique capable d’empêcher la bactérie de se multiplier. Pour la déterminer, il existe une méthode de référence appelée « méthode de dilution en milieu liquide ». Une gamme de tubes de bouillon de culture avec des concentrations croissantes de l’antibiotique à tester est préparée puis inoculée avec une même quantité de la bactérie étudiée. Après 18 heures d’incubation à 35°C, la concentration d’antibiotique la plus basse présente dans le tube pour lequel aucune croissance n’est observée (absence de trouble visuel du bouillon de culture) est considérée comme la CMI (Figure 2).
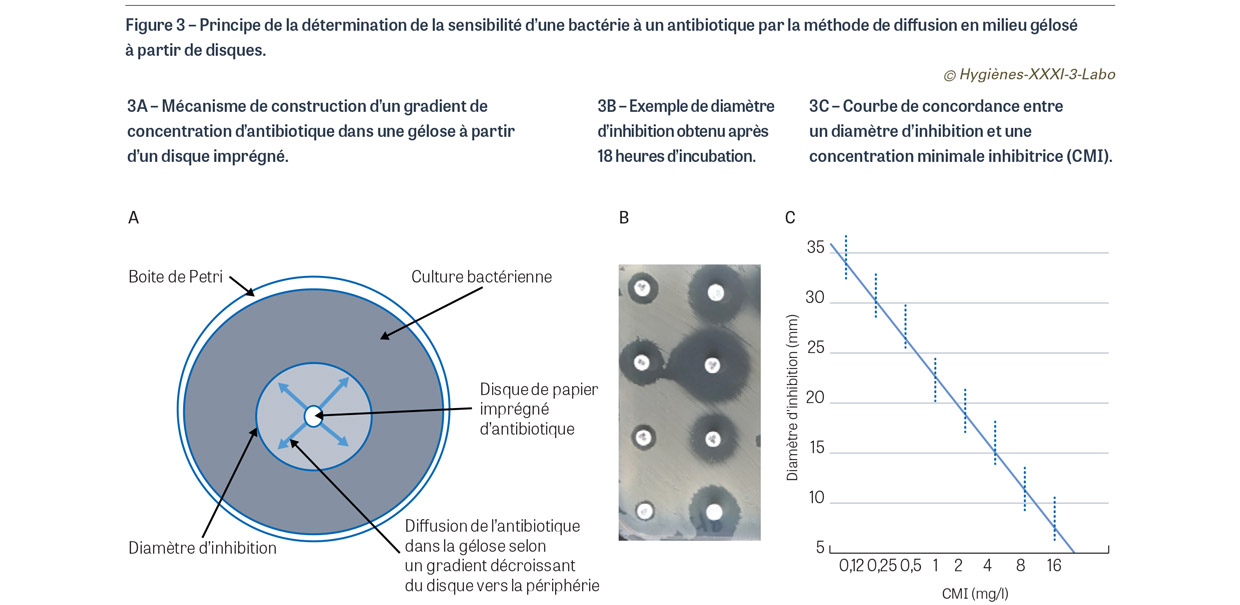
Comme cette technique est fastidieuse, les bactériologistes ont développé des méthodes plus simples qui permettent d’extrapoler cette CMI sans la mesurer directement. La plus répandue est la méthode de diffusion de concentrations connues d’antibiotique à partir de disques imprégnés : ces confettis calibrés sont posés sur une gélose pré-ensemencée par une suspension de concentration connue en bactéries pour lesquelles la CMI est à déterminer. Après 18 heures d’incubation, un diamètre d’inhibition est mesuré autour de chacun de ces disques : c’est la zone dans laquelle les bactéries présentes ne se sont pas développées car inhibées par l’antibiotique présent sur le disque. En effet l’antibiotique présent dans ce disque de cellulose a diffusé dans la gélose de manière graduellement décroissante en s’écartant du disque : plus on s’éloigne du centre du disque, plus la concentration en antibiotique présente dans la gélose diminue. Lorsque cette concentration diminue au-delà de la CMI spécifique de la bactérie testée, celle-ci est capable de se multiplier et de former des colonies visibles à l’œil nu. Il suffit alors de mesurer pour chaque antibiotique testé le diamètre d’inhibition et de le « traduire » en CMI par des courbes de concordance (abaques) réalisées par le fabricant de disques (Figure 3). Il existe également des méthodes automatisées qui permettent de simplifier la réalisation des antibiogrammes. Elles sont toujours basées sur l’évaluation des CMI des antibiotiques pour une souche bactérienne particulière.
Ces critères d’interprétation peuvent évoluer avec le temps, en fonction de la publication d’études d’efficacité chez l’homme ou de la description de l’apparition de nouvelles résistances qui s’expriment à bas niveau. Des études chez l’animal portant sur les paramètres pharmacocinétiques (absorption, diffusion, métabolisme et élimination de l’antibiotique dans l’organisme) et pharmacodynamiques (efficacité de l’antibiotique) des molécules associées à des approches mathématiques de modélisation viennent compléter et actualiser ces connaissances. Des stratégies sont également élaborées pour inciter les prescripteurs à utiliser les molécules et les schémas d’administration les moins susceptibles de favoriser l’émergence de résistances acquises.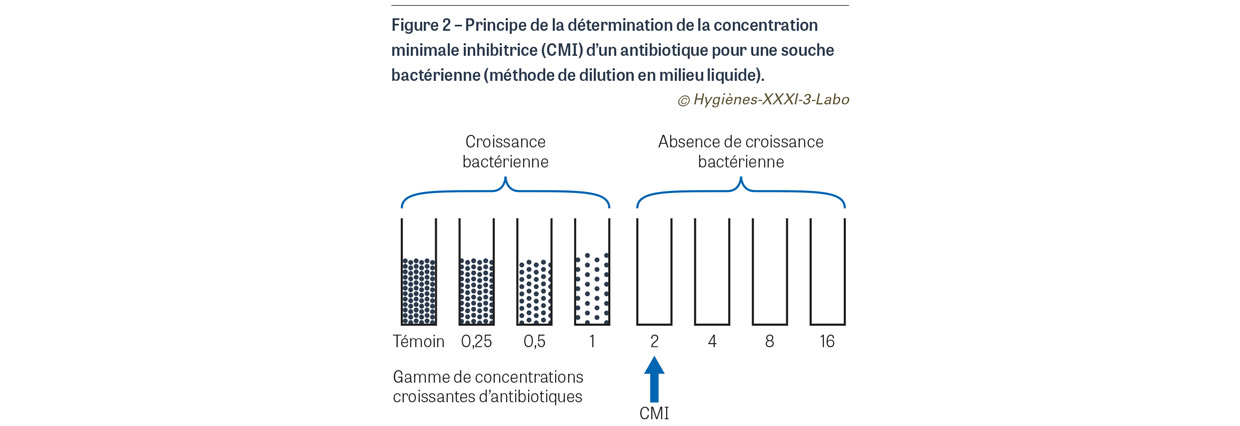
Comment interpréter un antibiogramme ?
Une fois la CMI d’un antibiotique mesurée ou extrapolée, il faut interpréter ce résultat et le classer dans une des trois catégories suivantes : si la souche y est sensible, l’utilisation de cet antibiotique à la dose standard préconisée par le résumé des caractéristiques du produit (RCP) aura une efficacité clinique dans au moins 90% des cas ; dans le cas d’une souche « intermédiaire » (dénommée maintenant « sensible à forte posologie », cf. ci-dessous), l’utilisation de cet antibiotique à forte dose aura une efficacité clinique dans au moins 90% des cas ; et si la souche est résistante à l’antibiotique, l’utilisation de celui-ci aura une probabilité d’échec clinique dans au moins 90% des cas quelle que soit la posologie. La définition de la catégorie « intermédiaire » a fait l’objet de modifications ces dernières années. Initialement, il était déconseillé d’utiliser un antibiotique classé comme « intermédiaire » ; cette catégorie était d’ailleurs regroupée avec la catégorie « résistante » dans les enquêtes épidémiologiques [1]. Depuis quelques années, la catégorie « intermédiaire » regroupe les souches bactériennes qui peuvent très bien être traitées par cet antibiotique à condition que celui-ci soit utilisé à forte dose [2]. À titre d’exemple, la posologie standard du céfépime pour les entérobactéries (E. coli, Klebsiella pneumoniae…) est de 1 g trois fois par jour alors que la forte posologie est de 2 g trois fois par jour, soit le double. Les souches classées « intermédiaires » sont désormais classées avec les souches « sensibles » dans les enquêtes épidémiologiques [1].
L’interprétation se fait grâce à des valeurs critiques qui correspondent aux valeurs maximales de concentration d’antibiotique atteignable dans le sang ou dans un autre site biologique concerné (urine, liquide céphalorachidien…) lors de son administration à la posologie standard ou maximale. Si la CMI est supérieure à cette concentration maximale d’antibiotique atteignable, cela signifie que l’antibiotique ne pourra pas inhiber la croissance de la bactérie et qu’il sera inefficace. D’autres méthodes de détermination de ces concentrations critiques existent, comme la méthode dite « épidémiologique » qui évalue la répartition des CMI d’un antibiotique pour un ensemble de souches, et l’identification d’une population majoritaire dite « sensible » et d’une population minoritaire devenue résistante, séparées par une valeur de concentration dite critique (Figure 4).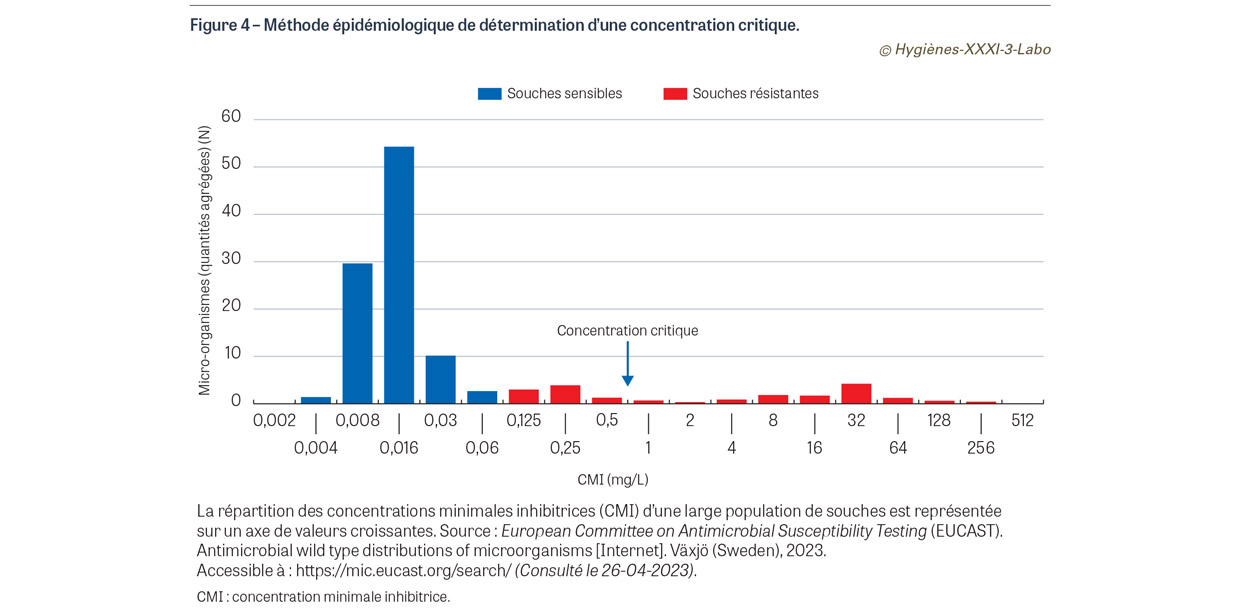
Limites de l’antibiogramme
Aujourd’hui, le principal défaut de l’antibiogramme est son délai de réalisation : il nécessite une culture pure de la bactérie, soit un premier délai de 18 à 24 heures, puis une incubation de même durée, soit un délai total au mieux de 48 heures. Alors que l’identification bactérienne a été raccourcie de 24 heures à quelques minutes par l’utilisation de la spectrométrie de masse, l’antibiogramme classique reste l’étape limitante d’une prise en charge rapide et ciblée des infections bactériennes. Dans certaines situations, le résultat de l’antibiogramme de première intention ne permet pas de s’assurer de la sensibilité de la souche bactérienne à l’antibiotique ou d’identifier la présence d’une résistance acquise. Il faut alors réaliser des tests complémentaires pour mettre en évidence des mécanismes particuliers de résistance ou déterminer précisément la CMI. Par ailleurs, la réalisation d’un antibiogramme par le laboratoire ne constitue pas une incitation à traiter et à prescrire des antibiotiques, hormis lorsqu’il porte sur une bactérie pathogène isolée d’un site stérile (sang circulant, liquide cérébro-spinal…). Le bon usage des antibiotiques inclut la juste prescription des tests diagnostiques (le diagnostic stewarship en anglais, qui vient compléter l’antimicrobial stewarship) : prescrits en excès, ils peuvent mettre en évidence des colonisations et contaminations qui n’ont aucun intérêt dans la prise en charge du patient mais augmentent inutilement la prescription d’antibiotiques, avec toutes les conséquences nocives à la fois individuelles et collectives, notamment sur la sélection et la diffusion de bactéries multirésistantes [6].
Conclusion
La résistance acquise aux antibiotiques existait au sein du monde bactérien avant la découverte et l’utilisation d’antibiotiques à large échelle par l’homme. Il est donc essentiel de pouvoir identifier très rapidement les antibiotiques utilisables pour traiter efficacement une infection sans trop perturber les microbiotes présents chez l’homme et dans l’environnement. L’antibiogramme est au cœur de cette stratégie et représente actuellement une cible privilégiée de nouvelles évolutions techniques, notamment pour améliorer son délai de rendu.
Note :
1- Acide désoxyribonucléique.